使用volume-defined ROI提取surface数据(比如, Cortial thickness)
来源:Freesurfer官方教程
https://surfer.nmr.mgh.harvard.edu/fswiki/VolumeRoiCorticalThickness
思路:把ROI转到surface空间
注意:tkmedit和tksurf似乎已经不支持64位,可以改用Freeview,命令不一样但不复杂。
材料:
1)一个Volume中定义的binary mask,把这个文件称为ROI5.nii。
2)个体(或者组平均)的T1 volume,需要和ROI在同一坐标空间。把这个文件称为TT_avg152T1.ni。
3) Freesurfer处理好的受试者(或一组受试者)的freesurfer数据。也就是说,recon-all -s <subj> -all已经运行完成,已有表面和皮质厚度数据。把这个被试称之为subjid。
流程:
1)画图确认下ROI5.nii中的ROI是不是在你想要的地方,这一步很多软件都可以轻易完成。

2)将ROI配准到freesurfer的模板上,推荐fsaverage。可以使用自动配准如fslregister或spmregister,或者手动配准如tkregister。其中Freesurfer提供的fslregister和spmregister分别使用的是FSL的flirt和SPM的spm_coreg,命令是一样的,这里使用了fslregister。
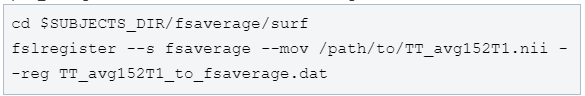
结果会生成一个TT_avg152T1_to_fssaverage.dat,它代表了从TT_avg152T1到fsaverage的变换矩阵。
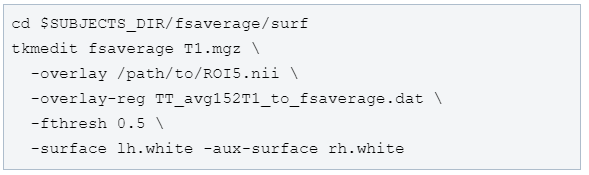
临时使用一下上面得到的变换矩阵,做个图看看是不是经过变换的ROI是不是想要的位置。
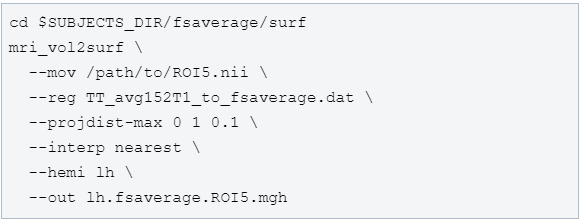
3)使用变换矩阵,将ROI变换到surface空间。
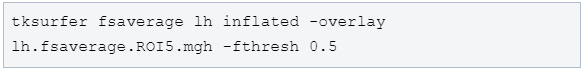
同样可以做个图看看变换的结果lh.fsaverage.ROI5.mgh是不是在想要的位置。
4)将个体空间的数据配到fsaverage标准空间中得到lh.thickness.fsaverage.mgh
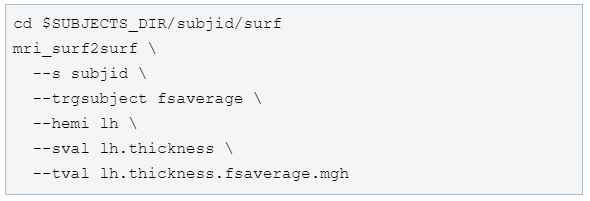
5)从lh.thickness.fsaverage.mgh中提取lh.fsaverage.ROI的数据。
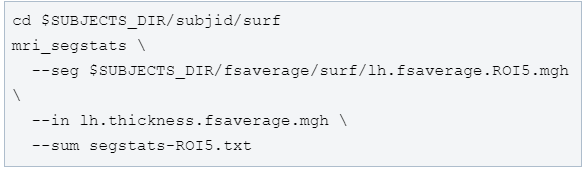
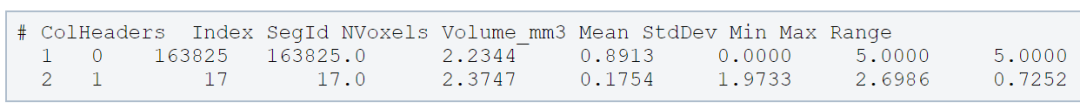
结果保存在txt中,1为ROI的皮层厚度2.3747。
使用surface-defined ROI/atlas提取Volume数据
思路:把某个ROI转到Volume空间。
来源:Andy的blog
https://andysbrainbook.readthedocs.io/en/latest/FreeSurfer/FS_ShortCourse/FS_11_ROIAnalysis.html
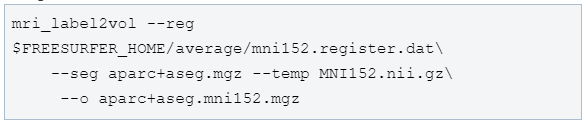
首先,用FreeSurfer的tkregister2命令创建一个名为register.dat的变换矩阵register.dat。其中beta_0001.nii是被试的Volume空间。

之后用mri_label2vol将其转到volume空间中,这里用到了之前得到的变换矩阵。
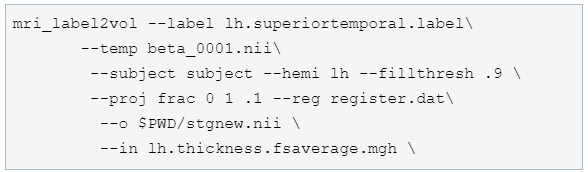
后续就是使用mask提取数据的常规操作了。
使用surface-defined atlas提取Volume数据
思路:将atlas转到volume空间。
来源:Freesurfer官网
https://surfer.nmr.mgh.harvard.edu/fswiki/CoordinateSystems
将DK模板转到MNI152空间,这里使用了Freesurfer自带的mni152的变换矩阵,因此不需要做配准。如果需要提取的数据在其他的空间,参见上面的tkregister2或者其他方式做配准获取相应的变换矩阵。
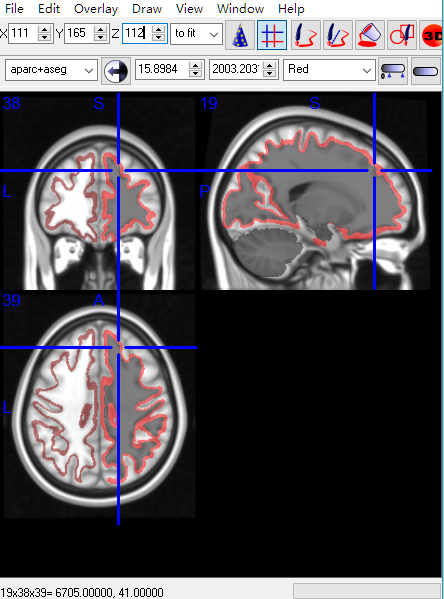
有个这个mask文件后,提取数据也是常规操作了。需要注意的是label的信息在Freesurfer中的FreeSurferColorLUT.txt文件中(如下图),这里mask的2026对应rh-rostralanteriorcingulate。

另外,mtrix中将Freesurfer默认的index和label做了调整,更直观。不熟悉mtrix用FS默认的即可。

https://mrtrix.readthedocs.io/en/latest/quantitative_structural_connectivity/labelconvert_tutorial.html
在FS的email list中有这样一个类似的问题,大概就是先找到皮层厚度差异的两个ROI,还想看一下这两个ROI的resting-state数据,怎么转到volume的空间。

得到的答复是这样的

直接把aparc+aseg.nii map到mni152_T1上
看起来是没问题的
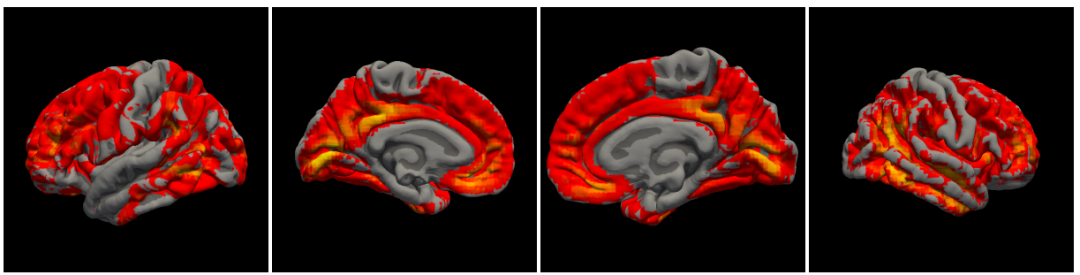
除了把ROI变换到数据空间,然后提取数据。是否可以把数据变换到ROI所在空间,进行提取呢?尝试了一下将MIN152空间的group level的数据变换到fsaverage空间,用的命令同样是mri_vol2surf。过程中如果出现voxel size的warnning可用3dresample可以解决。

不过效果一般,至少直接在group level上做是不好的。理想的做法是先在个体上做变换,然后得到在surface上操作得到group level的结果,应该就能得到比较好的coverage。
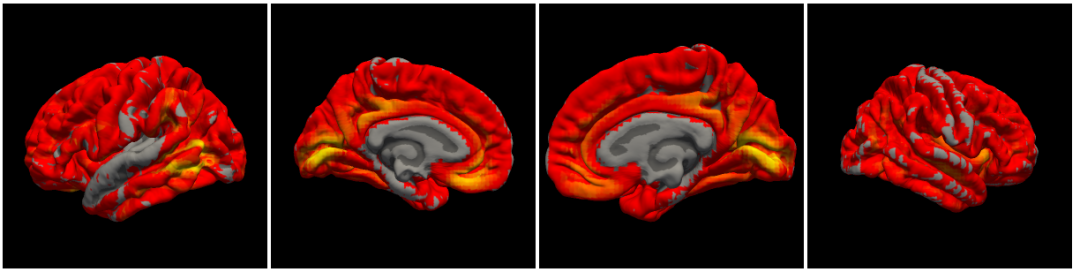
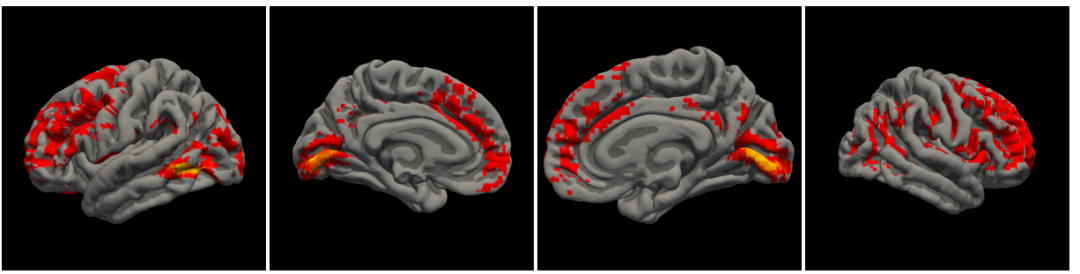
因此最后还是采用将DK模板转到volume空间的做法,之后根据index提取数据,需要注意voxel size和reslice的问题。
变换后的DK模板map到mni152_T1
由于做了resample所以分辨率在mni152上看有所下降
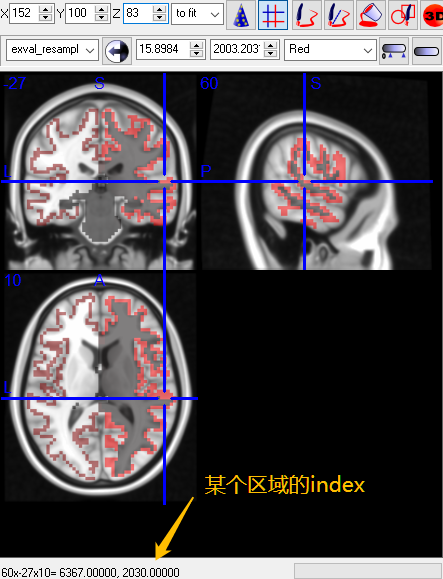
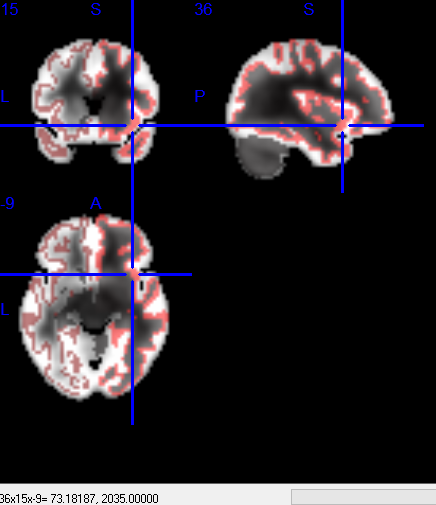
变换后的DK模板map到某个结果上
end







