超级计算机对科研工作的重要性不言而喻,强大的算力使得超算在几乎每个科研领域都做出了显著贡献。然而传统的超算实在是太!贵!!了!!!前期建设部署和后续使用维护都耗资不菲,周期也很长。

为解决这种问题,科学家们想出了很多方案。其中最有趣的,莫过于各种基于互联网的「分布式计算」,任何人只要下载一个客户端,即可在电脑闲置时自动运行来处理数据。例如斯坦福大学在二十多年前发起的Folding@Home项目,可将闲置算力汇集起来,用于精确模拟蛋白质折叠和错误折叠过程,以便更好地了解阿尔茨海默症、癌症等疾病的研究。类似的项目还有进行基因结构研究的Genome@Home等等。但时效性仍是问题,数据处理的完成时间难于预测。
在云计算迅猛发展的今天,科研工作者们是否有更好的方式,利用蛋白质折叠最新的开源成果,更快速地开展研究,并及时获得研究结果呢?
不妨试试「Folding@Azure」吧
Azure云提供的,即开即用的强大算力无疑是非常好的选择!我们不仅可以采用最新基于Milan CPU的超算机型,还有搭载Nvidia A100、V100、T4等丰富GPU加速计算的机型可以选择。本文将从详细的实操步骤出发,带领大家在Azure云上搭建RoseTTAFold运行环境。

阅读知晓RoseTTAFold及其权重数据的许可要求,并遵守条款中关于商业应用的约定;
注册申请PyRoseTTA的许可,通过后可下载对应版本的安装包(建议Python3.7 Linux版本);
注册开通Azure云的全球账号;
创建SSHKey并保存Pem私钥;
提交并完成Azure T4 GPU机型NCas_T4_v3的配额申请,若需更快性能,亦可使用搭载V100的NCs_v3系列;
该实验会产生费用,按T4机型一天完成来预估,在Southeast Asia区的计算及存储花费在$50以内,详细定价信息请参考这里,使用过程的账单数据可在控制台的订阅中查询。
确认上述准备事项完成后,让我们开动吧。
登录到Azure控制台,通过Home->Create Resource->Virtual Machine进入虚机创建页面,设置虚拟机基本参数(可参考下图)。
其中:虚机类型选择NC16as_T4_v3,操作系统镜像选择CentOS-based HPC 7.9 Gen2,该镜像已包含GPU驱动、CUDA工具及常用HPC套件,可以简化安装过程;SSH Key设为之前准备好的密钥。
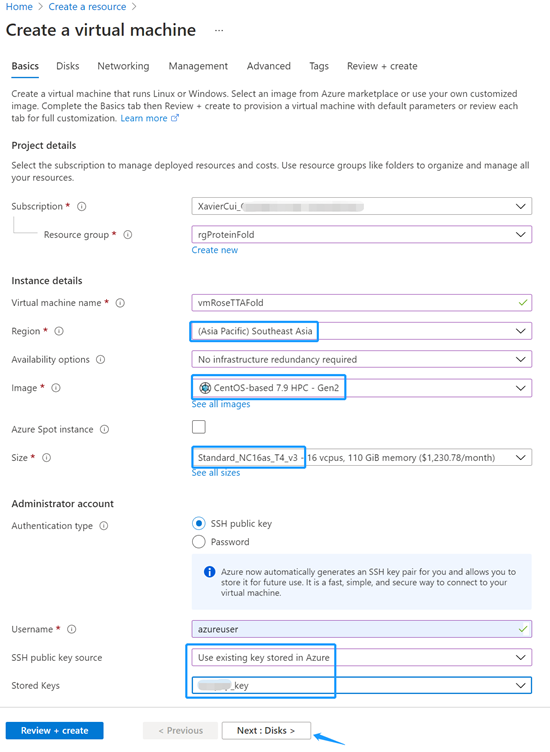
进入下一步,点击Create and attach a new disk来配置添加数据盘,考虑训练数据集的大小,建议至少配置为4096GB。点击Review + Create检查确认后点击Create创建。
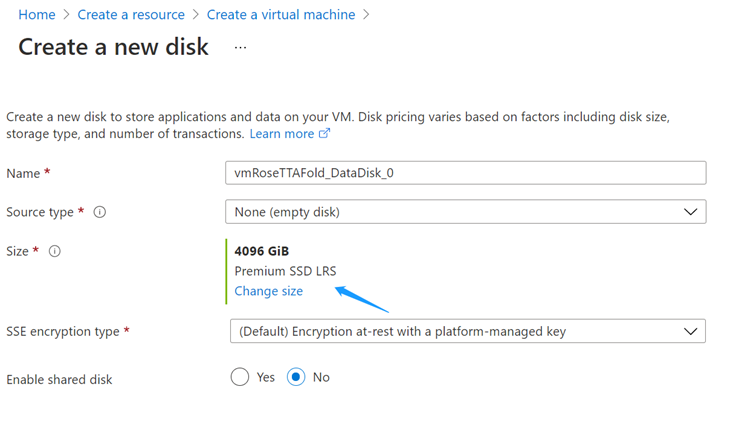
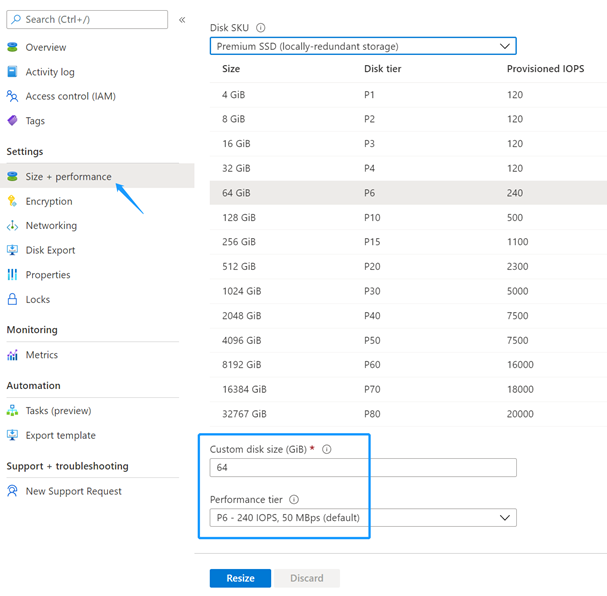
启动成功后还需执行一下系统盘扩容,操作顺序为:先停止虚机(操作时勾选「保留Public IP地址」),待虚拟机变成Stopped状态后,进入虚机的Disk->点选上面一栏系统盘->Size + Performance菜单配置系统盘大小为64G、性能为P6或更高,点击Resize待右上角状态提示成功后,回到虚机属性页Start操作,并等待几分钟待虚拟机状态变为Running就绪。
通过SSH登录到服务器,执行数据盘挂载操作如下。挂载成功后可通过lsblk命令检查/dev/sdc1磁盘状态:
sudo parted dev/sdc --script mklabel gpt mkpart xfspart xfs 0% 100%sudo mkfs.xfs dev/sdc1sudo partprobe dev/sdc1sudo mkdir datasudo mount dev/sdc1 datasudo chown azureuser:azureuser data/uuidstr=$(blkid | grep /dev/sdc1 | awk -F " " '{print $2}' | awk -F= '{print $2}' | sed 's/"//g')cat <<EOF | sudo tee -a etc/fstabUUID=$uuidstr data xfs defaults, nofail 1 2EOFlsblk -o NAME,HCTL,SIZE,MOUNTPOINT | grep -i "sd"
虚机配置就绪后,在SSH登录中继续执行以下安装指令,这些指令包含:
安装Anaconda3,安装步骤中阅读License空格翻页,安装路径指定为/opt/anaconda3,询问是否Conda初始化时选Yes;
下载RoseTTAFold Github资源;
配置Conda env环境;
在Folding环境中安装核心的PyRosetta4组件。
## Install anacondawget https://repo.anaconda.com/archive/Anaconda3-2021.05-Linux-x86_64.shchmod +x Anaconda3-2021.05-Linux-x86_64.shsudo bash ./Anaconda3-2021.05-Linux-x86_64.shcat <<EOF | sudo tee -a etc/profileexport PATH=\$PATH:/opt/anaconda3/binEOFsource /etc/profile## Get repo and buildcd /datagit clone https://github.com/RosettaCommons/RoseTTAFold.gitcd RoseTTAFoldconda env create -f RoseTTAFold-linux.ymlconda env create -f folding-linux.ymlconda env list./install_dependencies.shconda init bashsource /home/azureuser/.bashrcconda activate foldingwget https://asiahpcgbb.blob.core.windows.net/rosettaonazure/PyRosetta4.Release.python37.linux.release-289.tar.bz2tar -vjxf PyRosetta4.Release.python37.linux.release-289.tar.bz2cd PyRosetta4.Release.python37.linux.release-289/setuppython setup.py installpython #To verify the pyrosetta lib in python commandline#then input two commands: <<<import pyrosetta; <<<pyrosetta.init()conda deactivate #back to conda (base)conda deactivate #back to VM shell
(folding) [azureuser@vmRoseTTAFold RoseTTAFold]$ pythonPython 3.7.10 (default, Jun 4 2021, 14:48:32)[GCC 7.5.0] :: Anaconda, Inc. on linuxType "help", "copyright", "credits" or "license" for more information.>>> import pyrosetta;>>> pyrosetta.init()PyRosetta-4 2021 [Rosetta PyRosetta4.Release.python37.linux 2021.27+release.7ce64884a77d606b7b667c363527acc846541030 2021-07-09T18:10:05] retrieved from: http://www.pyrosetta.org(C) Copyright Rosetta Commons Member Institutions. Created in JHU by Sergey Lyskov and PyRosetta Team.core.init: Checking for fconfig files in pwd and ./rosetta/flagscore.init: Rosetta version: PyRosetta4.Release.python37.linux r289 2021.27+release.7ce6488 7ce64884a77d606b7b667c363527acc846541030 http://www.pyrosetta.org 2021-07-09T18:10:05core.init: command: PyRosetta -ex1 -ex2aro -database /home/azureuser/.conda/envs/folding/lib/python3.7/site-packages/pyrosetta-2021.27+release.7ce6488-py3.7-linux-x86_64.egg/pyrosetta/databasebasic.random.init_random_generator: 'RNG device' seed mode, using '/dev/urandom', seed=-2055557650 seed_offset=0 real_seed=-2055557650basic.random.init_random_generator: RandomGenerator:init: Normal mode, seed=-2055557650 RG_type=mt19937>>>
按Ctrl+D退出,并执行conda deactivate返回至虚拟机环境。
紧接着是数据准备,包含权重数据和参考蛋白质样本库。数据量较大,我们在Azure Blob上存放了这些数据的副本,建议通过这些链接下载速度更快。由于有些单体数据大,下载后解压会比较慢(小时级),可以打开登陆多个SSH进程分别执行且勿中断保证数据完整。
解压完成后可用du -sh <dirname>命令检查bfd、pdb100和UniRef30三个目录的数据量分别应为1.8TB、667GB和181GB。
cd /data/RoseTTAFold/## wget https://files.ipd.uw.edu/pub/RoseTTAFold/weights.tar.gzwget https://asiahpcgbb.blob.core.windows.net/rosettaonazure/weights.tar.gztar -zxvf weights.tar.gz## uniref30 [46G]## wget http://wwwuser.gwdg.de/~compbiol/uniclust/2020_06/UniRef30_2020_06_hhsuite.tar.gzwget https://asiahpcgbb.blob.core.windows.net/rosettaonazure/UniRef30_2020_06_hhsuite.tar.gzmkdir -p UniRef30_2020_06tar -zxvf UniRef30_2020_06_hhsuite.tar.gz -C ./UniRef30_2020_06## BFD [272G]## wget https://bfd.mmseqs.com/bfd_metaclust_clu_complete_id30_c90_final_seq.sorted_opt.tar.gzwget https://asiahpcgbb.blob.core.windows.net/rosettaonazure/bfd_metaclust_clu_complete_id30_c90_final_seq.sorted_opt.tar.gzmkdir -p bfdtar -zxvf bfd_metaclust_clu_complete_id30_c90_final_seq.sorted_opt.tar.gz -C ./bfd## structure templates (including *_a3m.ffdata, *_a3m.ffindex) [over 100G]## wget https://files.ipd.uw.edu/pub/RoseTTAFold/pdb100_2021Mar03.tar.gzwget https://asiahpcgbb.blob.core.windows.net/rosettaonazure/pdb100_2021Mar03.tar.gztar -zxvf pdb100_2021Mar03.tar.gz
接下来即可运行RoseTTAFold应用了:(注意脚本第二个参数勿遗漏,它代表当前工作路径)。
cd example../run_pyrosetta_ver.sh input.fa .
首次运行需构建MSA参数及执行HHsearch搜索,时间较长,建模及预测约需30分钟,执行成功的输出如下,example/model/路径下会生成优选的5个pdb蛋白质模型结果命名为model_x.pdb,AI模型训练的过程日志可在./log/folding.stdout文件查看。
[azureuser@vmRoseTTAFold example]$ ../run_pyrosetta_ver.sh input.fa .Running HHblitsRunning PSIPREDRunning hhsearchPredicting distance and orientationsRunning parallel RosettaTR.pyRunning DeepAccNet-msaPicking final modelsFinal models saved in: ./modelDone
mv t000_.3track.npz t000_.3track.npz.origin../run_e2e_ver.sh input.fa .
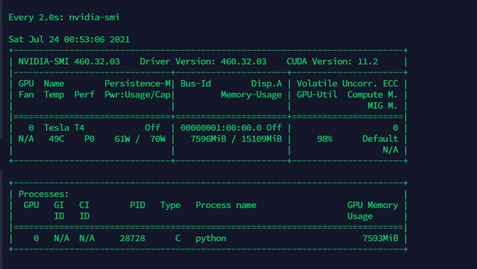
在预测步骤时可观察到GPU利用率接近满载,截图如下所示。另外,run_pyrosetta_ver.sh和run_e2e_ver.sh中(代码第18行)的CPU和内存都默认设为8 vCPU及64G RAM,如需更多的CPU资源加入或使用Azure更高配置机型时,运行前可手动修改这一配置以充分利用计算资源。
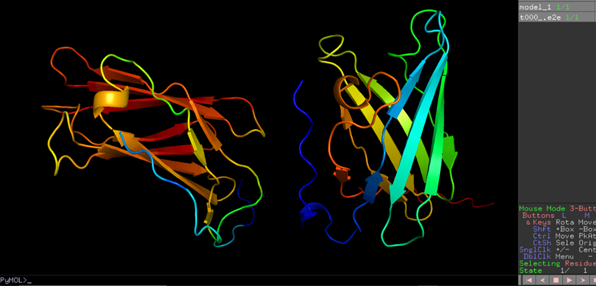
计算完成后,在PyMOL工具中查看分别用pyrosetta和end2end生成的两个pdb蛋白质结构如下图所示。接下来就是把输入换成您的fasta文件了,还可以根据这两个脚本的示范来编写您自己的运行脚本。
总结
以上就是单个虚机上搭建蛋白质折叠分析环境的过程,虚机在不使用的情况可将其停止以节约费用,数据盘建议生成快照进行备份,后续可随时重建。蛋白质样本库还可用AZcopy工具上传存入您账号下的Blob存储中以备后用。在确认不再使用计算和存储时,控制台中删除资源组即可清除对应资源。
有问题可通过GitHub反馈或讨论。对于大规模的并行分析,我们亦有构建HPC版RoseTTAFold高性能集群的解决方案,欢迎与我们联系对接。
希望Azure云计算助力加速您星辰大海的征途!

动手实验中用到的VM镜像名称是?
1


点击“在看”,分享Azure 云资讯