介绍
预训练的Med3D为之后的医学分割检测任务带来了很大的优势。这个预训练的模型,就像计算机视觉中常用的BackBone,可以用来初始化自己的任务,进行迁移学习,相比于从头训练,精度提升范围在3%-20%。
目前Med3D的模型已经集成在Onekey平台,相关的材料可以关注公众号,后台回复「Med3D」获取。另外可以加入QQ群参与讨论:537345968
模型的训练方法在论文Med3D: Transfer Learning for 3D Medical Image Analysis中进行了详细描述,主要包含以下几点贡献:
将多个医学比赛中的数据进行了整合,创建了3DSeg-8数据集,数据中包含不同的模态、不同目标器官和不同的病理情况。
设计了一个3D网络(Med3D),共同训练多种训练数据,产生了3D-ResNet系列预训练模型,可作为backbone用于分割、检测和分类任务。
使用训练好的预训练模型,对多个分割任务进行了测试,通过使用该预训练模型进行迁移学习,相比于从头训练,精度提升范围在3%-20%。
网络训练方法
数据选择
8个分割数据集,来源于不同的模态(MRI和CT),不同的扫描区域(脑、心脏、胰腺、脾脏等),3D中不同的分辨率、不同的像素灰度范围。
数据预处理:空间和灰度分布归一化。
空间归一化:spacing归一化。为防止过插值,对面每一个体素的插值,目标spacing选为该体素对应的区域内的所有体素的中值spacing,以保证体素内目标不会成为过插值而丢失其本身的特征。
灰度归一化:不同图像模态灰度范围是不相同的。需要对灰度做归一化。同时,为了消除离群点(异常点,灰度很高或很低的点),对灰度范围0.5%-99.5%以外的做一个截断。然后采用减去均值,和除以标准差的形式,对矫正后的灰度图像进行归一化。
Med3D网络结构
采用ResNet网络为基本结构。
存在的问题是,虽然不同的数据集中都有多个组织,但是每个数据集只有一个组织的分割Mask(如肝脏分割数据集,肯定只有肝脏的Mask,虽然里面也许存在胰腺,但是胰腺的label)。直接拿这些数据集进行训练,会出现问题,导致网络难以收敛。
训练方法:
如图1,训练方法不是将8个数据集混在一起,去做训练。而是以一种并联的形式,用同一个Bacbone(encoder)结构,后面并联接8个分支的decoder结构(红色块),每个分支针对一种数据集。所以尽管encoder共享了,但是decoder是不同的,就可以使网络在decoder上去适应不同的分割目标,而encoder就成了一个通用的结构,具有普适性。
另外网络中八个分支的decoder都是很简单的结构,会直接upsample后,和ground truth计算Loss,这样一个简单的decoder更有助于encoder储藏更多信息,且学到更多普适的信息。
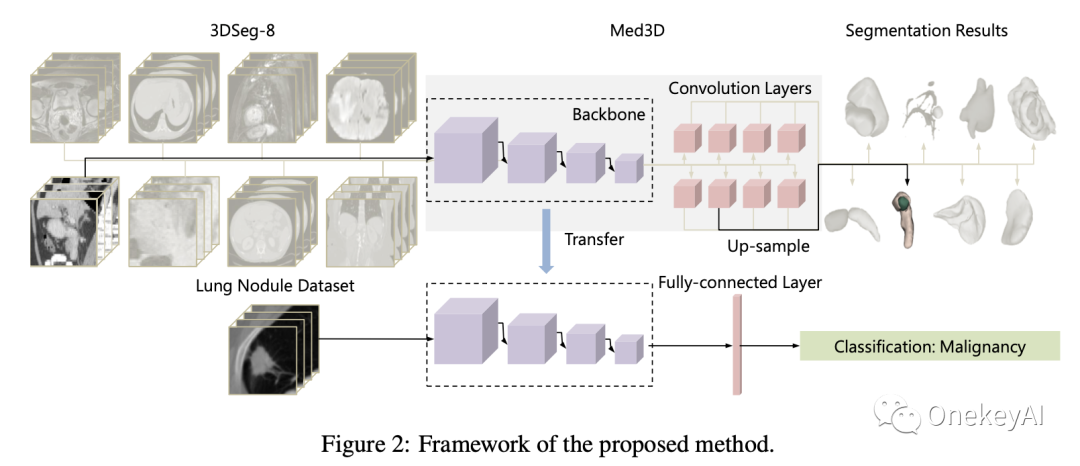
图1 Med3D训练方法
迁移学习效果
作者训练出了一个Backbone,这个可用于分类、检测、分割等。
作者把backbone用到下面图2这个网络结构,用于肝脏分割比赛LiTS数据集做测试,效果接近于使用多模型集成的方法,比单一网络的方法效果都要好。
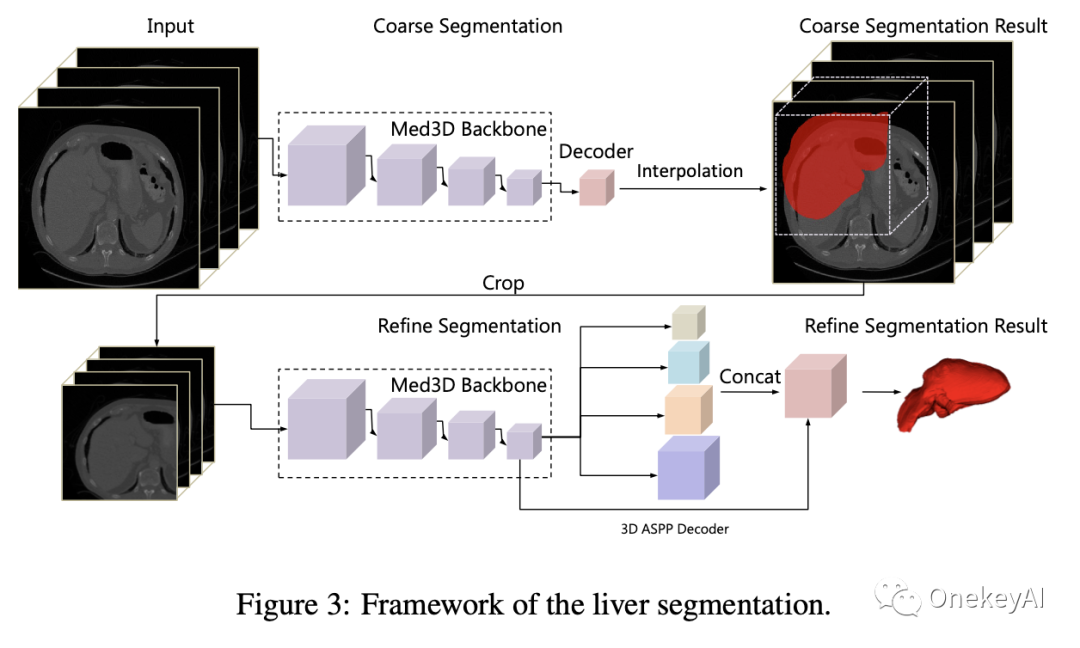
图2 肝脏分割框架
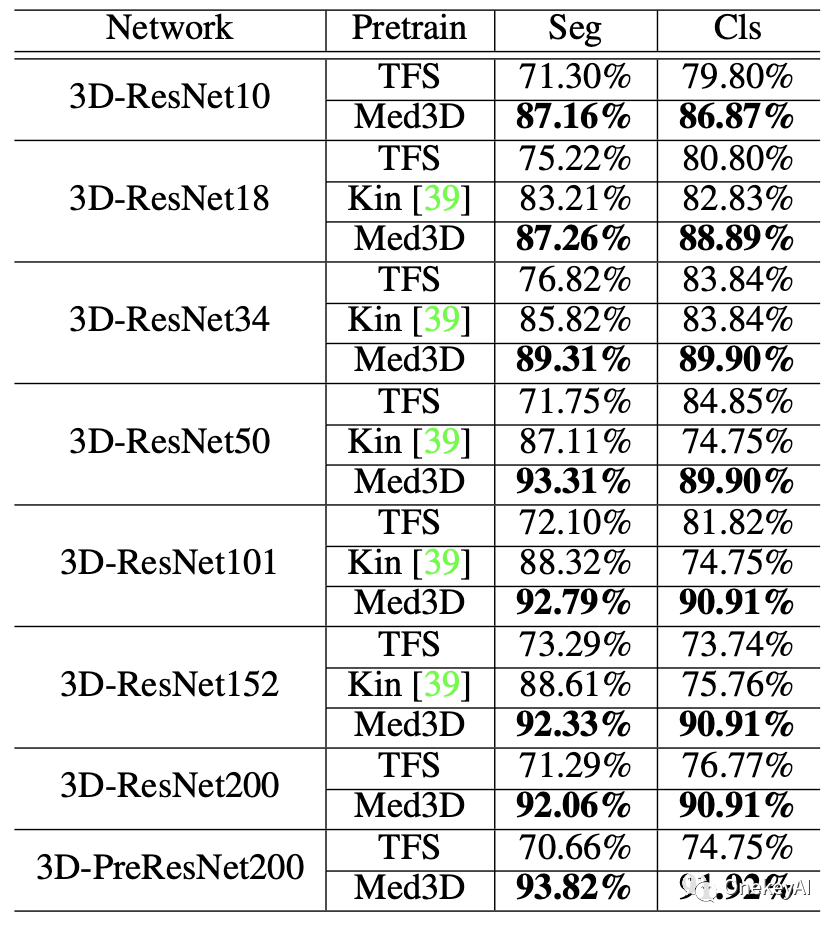
图3 使用预训练模型和从头训练效果对比
图3是作者将迁移学习应用到肺分割和肺结节分类,和从头训练的精度的对比。Med3D比Kin和TFS都要好,提升效果非常明显。分割和分类任务,Med3D都完美胜任。从头训练的网络随着网络结构加深,会出现过拟合,而Med3D随着网络加深,精度会逐渐提升,完全没有过拟合问题。
关注「OnekeyAI」公众号,后台回复「Med3D」获取Med3D代码、模型等更多资料。

好消息:BraTS相关代码、数据已经集成在Onekey平台中,如需购买「长按QR码识别」,案例关注公众号购买。







