昨天转载了一篇公众号推文,简单粗暴的了解测序技术。目前使用最为广泛的还是二代测序技术。如果有小伙伴提前了解过这方面的知识的话,你刚开始是不是会好奇(为什么一个个体为什么会有两个文件呢,xxx.fastq1、xxx.fastq2?)当时我也不是很懂测序的原理,就去问了我的同门,他说:测序的时候一个片段,正向测一次,反向再次一起,当时说我的一脸懵逼,后来看了 Illumina 公司的测序原理我才恍然大悟。
B 站Illumina 测序原理视频:https://www.bilibili.com/video/BV1ht411q7Wh?from=search&seid=9937739623014885354
了解质控
当我们拿到数据后,需要对数据进行质量控制(质控),这是因为一般测序下机数据会存在含N比例过大、测序质量较低的碱基数占比过高、含有duplication、序列污染等低质量reads,这些不合格的reads会影响后续的分析。对于质控我们主要了解的内容包括含N比例、GC含量、duplication情况、序列长度分布情况、碱基平衡情况等。今天,我们将一起通过数据格式和质量体系、数据质控步骤、Fastqc结果解读及异常处理三大模块进行学习。
Fastq文件格式说明
| FASTQ文件每个序列通常为4行,分别为: |
|---|
| Line 1 begins with a '@' character and is followed by a sequence identifier and an optional description (like a FASTA title line). |
| Line 2 is the raw sequence letters. |
| Line 3 begins with a '+' character and is optionally followed by the same sequence identifier (and any description) again. |
| Line 4 encodes the quality values for the sequence in Line 2, and must contain the same number of symbols as letters in the sequence. |
FASTQ的文件示例:
@DJB775P1:248:D0MDGACXX:7:1202:12362:49613 1:Y:18:ATCACGTGCTTACTCTGCGTTGATACCACTGCTTAGATCGGAAGAGCACACGTCTGAA+JJJJJIIJJJJJJHIHHHGHFFFFFFCEEEEEDBD?DDDDDDBDDDABDDCA
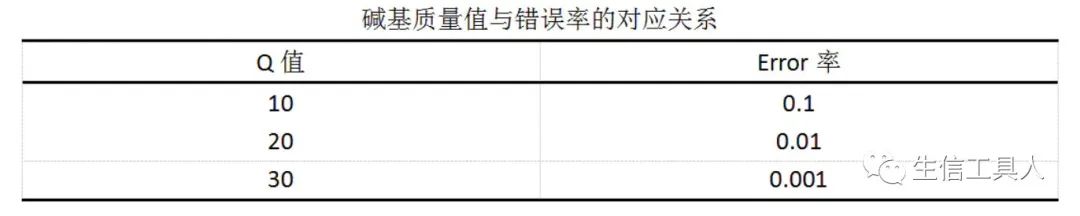 即,Q10准确率为90%,Q20准确率为99%,Q30准确率为99.9%,Q40准确率为99.99%,Q50准确率为99.999%。
即,Q10准确率为90%,Q20准确率为99%,Q30准确率为99.9%,Q40准确率为99.99%,Q50准确率为99.999%。FastQC质量报告
质量控制的软件很多,但是目前主要以fastqc为主。常见的用法:
FastQC分析压缩的fastq文件
fastqc -o xx/yy --noextract -f fastq -t 4 Pseudosciaena-A987-T01_good_1.fq.gz Pseudosciaena-A987-T01_good_2.fq.gz
开始分析:
 结果会得到一个html文件和一个zip压缩包
结果会得到一个html文件和一个zip压缩包 其中html文件用浏览器打开就能直观看到数据。
其中html文件用浏览器打开就能直观看到数据。报告图
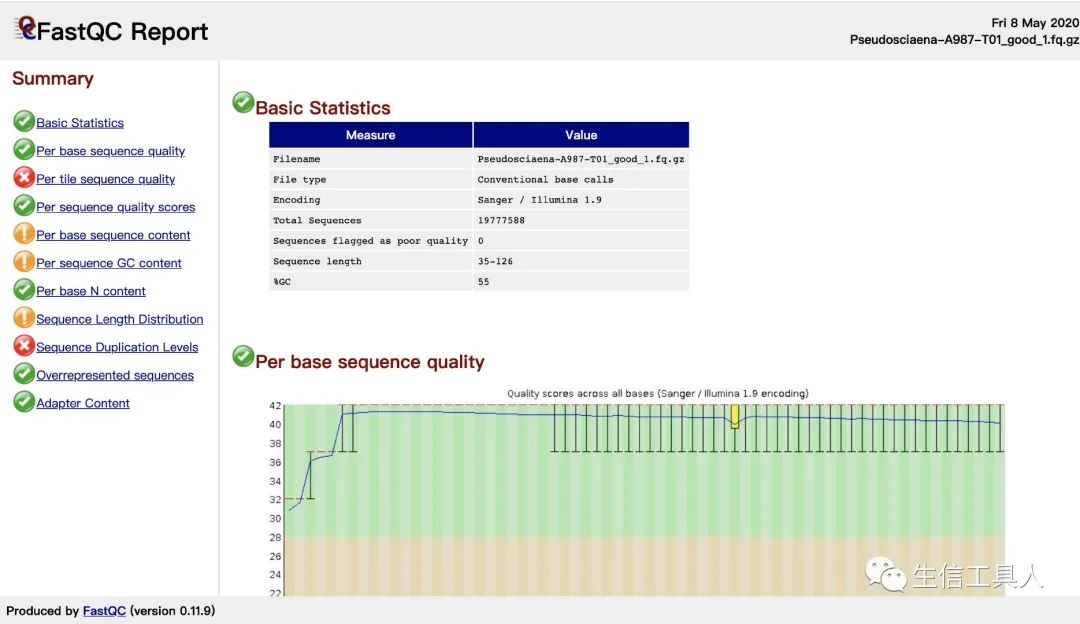
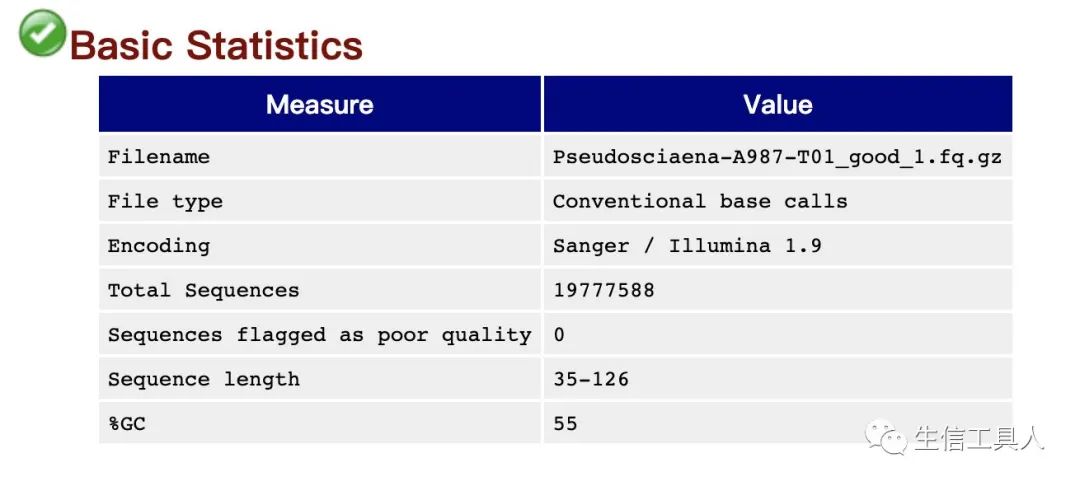
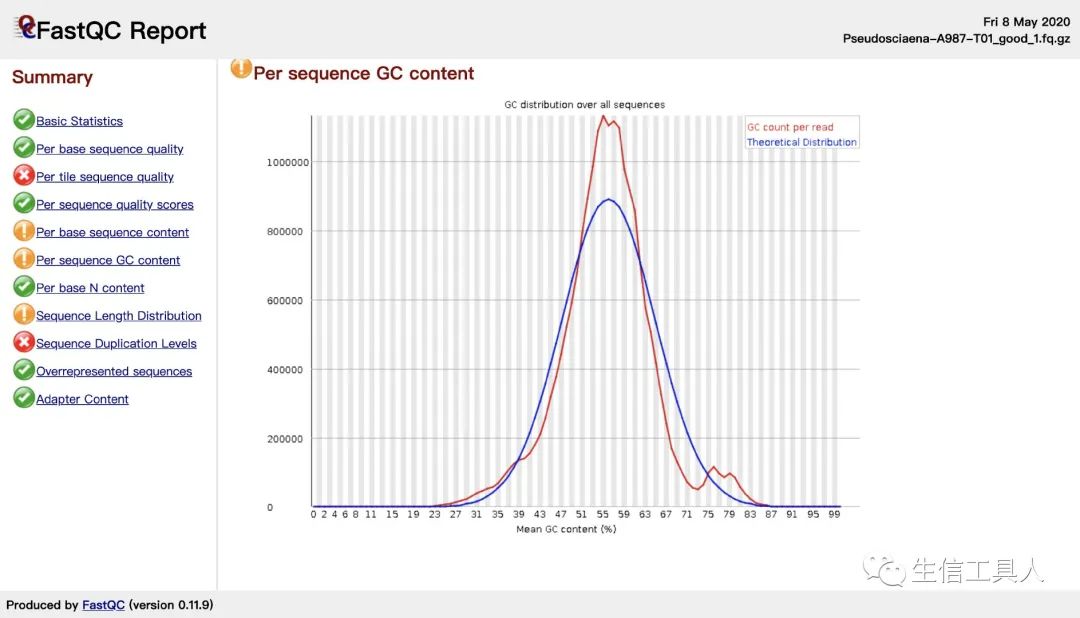
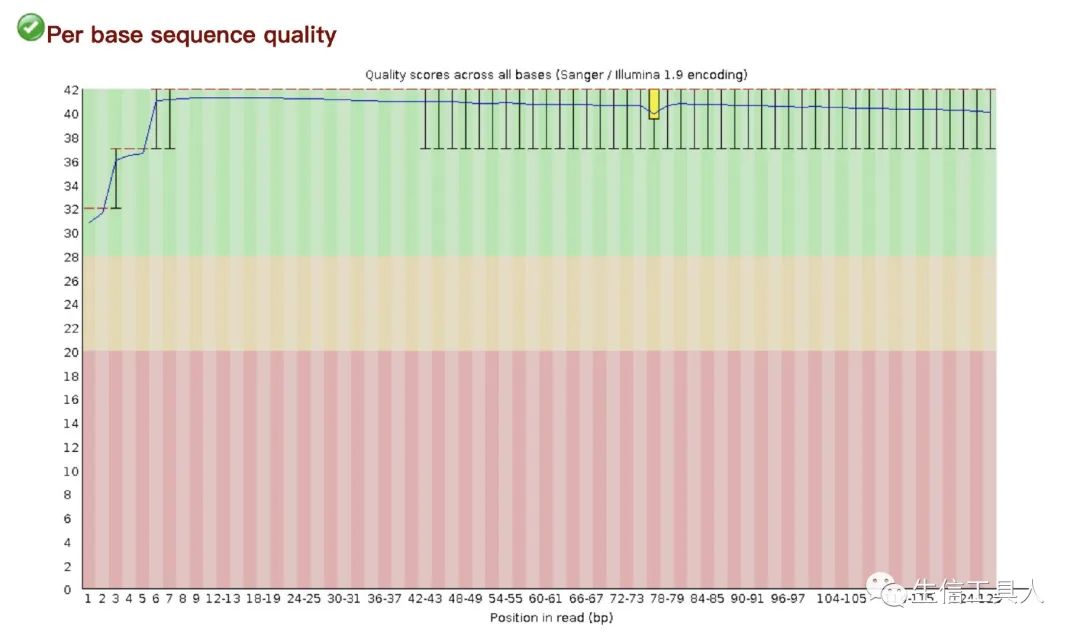 第一张图的左侧显示了一个 fastq 文件的质控目录第二张图为基本统计,第三张图为每个碱基的质量。箱线图中,红色表示中位数,黄色是25%-75%区间,触须是10%-90%区间,蓝线是平均数。若任一位置的下四分位数低于10或中位数低于25,报"WARN";若任一位置的下四分位数低于5或中位数低于20,报"FAIL"。当出现任一位置的下四分位数低于10或中位数低于25或任一位置的下四分位数低于5或中位数低于20时,表示测序数据存在质量不合格的情况,这时我们可以继续观察Sequence Content 图、GC Content 图、N Content 图 、Sequences Duplication level 图,这几个图进一步判断测序数据的不合格之处具体在哪。观察Sequence Content图和GC Contenta图的GC含量的线是否平行于X轴,若不平行,则该位置往往有over represented sequence的污染,可能原因建库过程的误差、测序的系统误差或者文库本身特点。
第一张图的左侧显示了一个 fastq 文件的质控目录第二张图为基本统计,第三张图为每个碱基的质量。箱线图中,红色表示中位数,黄色是25%-75%区间,触须是10%-90%区间,蓝线是平均数。若任一位置的下四分位数低于10或中位数低于25,报"WARN";若任一位置的下四分位数低于5或中位数低于20,报"FAIL"。当出现任一位置的下四分位数低于10或中位数低于25或任一位置的下四分位数低于5或中位数低于20时,表示测序数据存在质量不合格的情况,这时我们可以继续观察Sequence Content 图、GC Content 图、N Content 图 、Sequences Duplication level 图,这几个图进一步判断测序数据的不合格之处具体在哪。观察Sequence Content图和GC Contenta图的GC含量的线是否平行于X轴,若不平行,则该位置往往有over represented sequence的污染,可能原因建库过程的误差、测序的系统误差或者文库本身特点。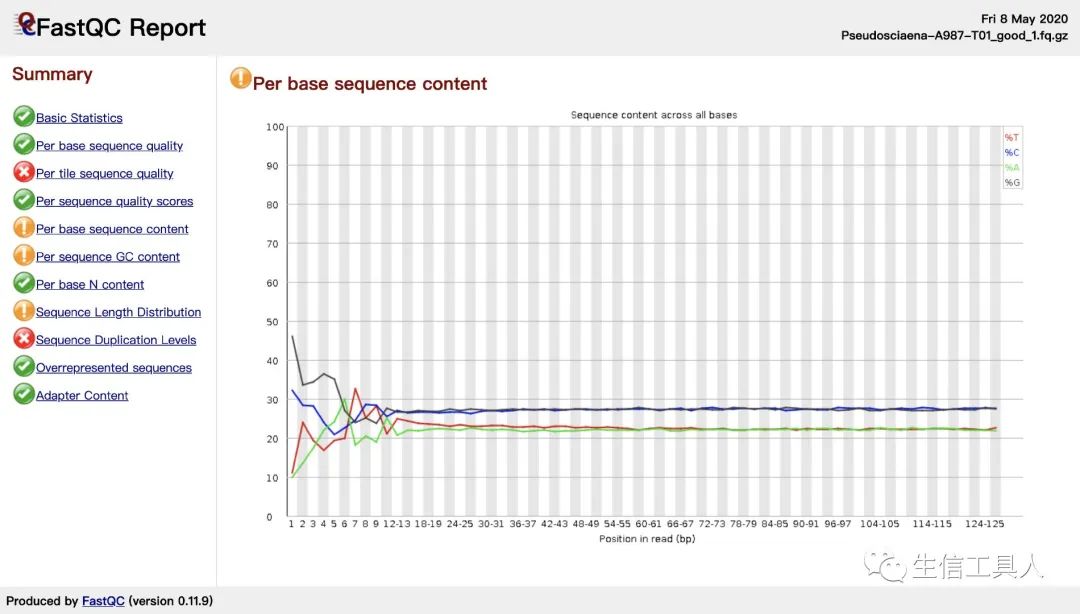
 由N Content 图可知reads中含N碱基的情况,理想状况下是含N量越少越好,在微生物多样性分析中一般是去除含N碱基比例>5%的序列。
由N Content 图可知reads中含N碱基的情况,理想状况下是含N量越少越好,在微生物多样性分析中一般是去除含N碱基比例>5%的序列。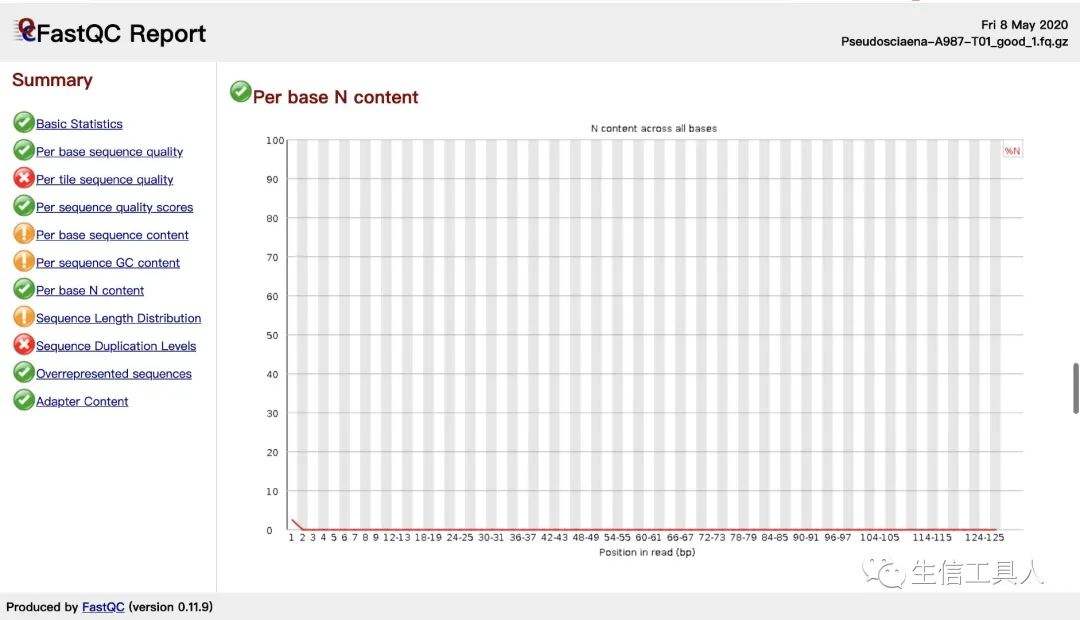 观察Sequences Duplication level 图,横坐标是duplication的次数,纵坐标是duplicated reads的数目,若duplication的程度偏高,则可能存在PCR duplication。去除duplication可以通过Samtools、Picard或Iontorrent,其中Samtools只看5’端的起始位置不考虑reads突变;Picard不仅考虑起始位点也会考虑突变情况和质量值,即reads完全一样的才会被当成duplication被去除;Iontorrent则是看5’端的起始位置和3’端adaptor的比对情况,不考虑reads突变。
观察Sequences Duplication level 图,横坐标是duplication的次数,纵坐标是duplicated reads的数目,若duplication的程度偏高,则可能存在PCR duplication。去除duplication可以通过Samtools、Picard或Iontorrent,其中Samtools只看5’端的起始位置不考虑reads突变;Picard不仅考虑起始位点也会考虑突变情况和质量值,即reads完全一样的才会被当成duplication被去除;Iontorrent则是看5’端的起始位置和3’端adaptor的比对情况,不考虑reads突变。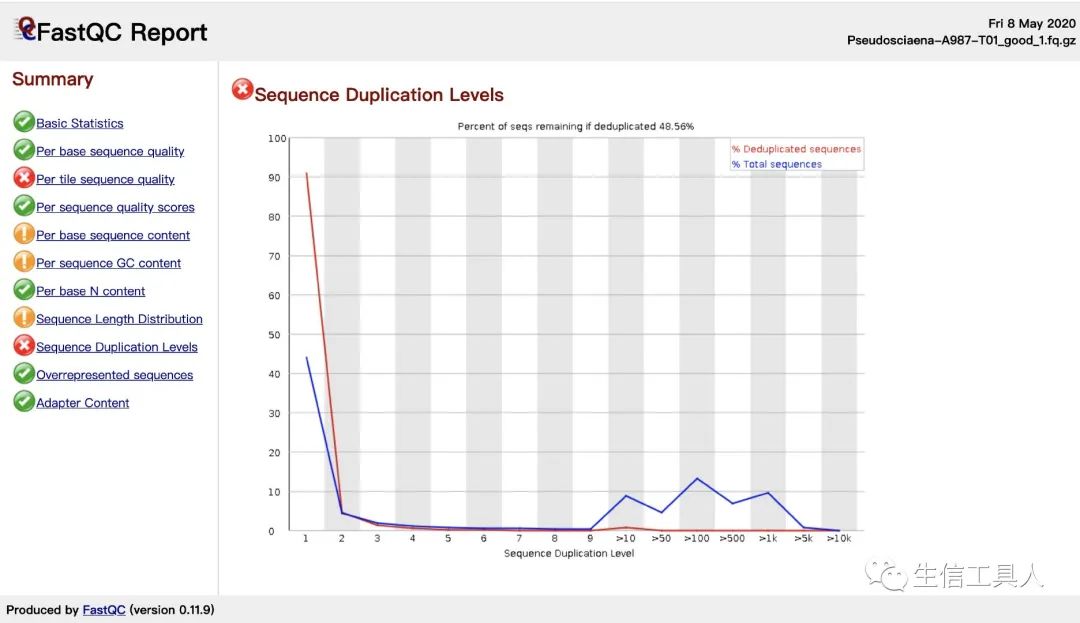 最后,在进行去低质量reads和接头等预处理步骤后,再次进行fastqc质控。
最后,在进行去低质量reads和接头等预处理步骤后,再次进行fastqc质控。
结束语
今天主要讲的质控的步骤和图的简单理解,搜狐有一篇比较详细的 fastqc 结果的解读(网址:https://www.sohu.com/a/316999025_769248)。关于质控文件出现问题时如何解决,且看下回分解!我还是那个在生信路上摸爬滚打的工具人。一起加油吧!如果需要提供帮助或者是反馈意见,可以发送邮件到 liwei12306@163.com







