点击上方↗️「活水智能」,关注 + 星标🌟

作者:Giuseppe Futia
编译:活水智能
随着科学研究的蓬勃发展、技术的不断进步以及全球对医疗健康研究的重视,生物医学领域的出版率多年来稳步上升。
自然语言处理(NLP)技术在生物医学领域的应用,标志着对庞大生物医学知识语料进行分析和解读的方式发生了转变,从文本数据中提取有意义洞察的能力得到了显著提升。
命名实体消歧(Named Entity Disambiguation, NED)是 NLP 中的一项关键任务,旨在通过将实体提及链接到知识库中的正确条目来解决歧义问题。为了理解这一任务的重要性和复杂性,可以考虑以下示例:
Zika 属于黄病毒科,通过埃及伊蚊传播。感染 Zika 的个体通常会出现发热、关节痛和神经系统并发症等症状。Zika 的严重后果是由于其在怀孕期间能够穿过胎盘屏障,导致小头畸形和胎儿畸形。
这段文字中的三个句子表面上提到了同一个由术语“Zika”表示的“事物”。然而,通过分析每个句子中的上下文词汇和实体,可以发现:
Zika 属于 黄病毒科,通过 埃及伊蚊 传播。感染 Zika 的个体通常会出现诸如 发热、关节痛 和 神经系统并发症 等症状。Zika 的严重后果是由于其在 怀孕 期间能够穿过 胎盘屏障,导致 小头畸形 和 胎儿畸形。
通过分析每个句子中的实体,可以清楚地看到,“Zika” 的每次提及可能都指代了不同的现实世界实体。例如,在第一句中,“Zika” 很可能指代病毒,因为句中提到了病毒所属的科目以及传播媒介。在第二次提及中,“Zika” 可能指代由 Zika 病毒引起的疾病,因为提到了多种症状。最后一次提及则与影响胎儿的特定 Zika 形式相关。
这种隐含的 Zika 相关实体的区分可以通过知识库(如统一医学语言系统 UMLS)显式表示,该知识库提供了关于“Zika 病毒”、“Zika 病毒感染”和“先天性 Zika 综合征”的以下信息:
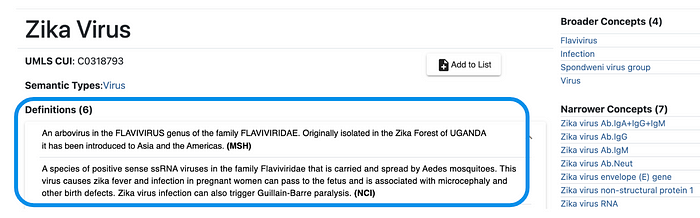
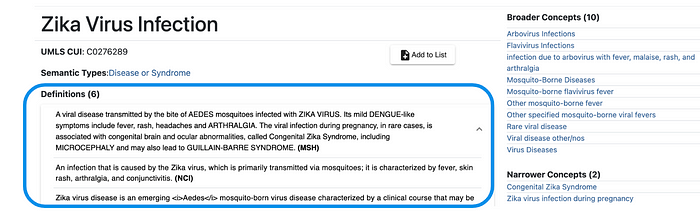
如上述示例所示,每个实体均与多段文本相关联。这些文本信息可以转化为一组特征,用于包括命名实体消歧在内的各种机器学习任务。然而,这些信息在知识库中的分布并不一致。例如,“先天性 Zika 综合征”缺乏定义,而其他实体则提供了定义。因此,我们需要能够利用超越文本信号的其他信息来源的方法。
提出的方法
如前文所述,传统的 NED 方法主要依赖于从知识库中提取的文本内容。虽然这些方法充分利用了这些信息,但它们可能缺乏从目标实体与知识库中其他实体之间的关联中获得的全面关系洞察。这一点在文本信息缺失(尤其是对于较少见的实体)时表现得尤为明显。
NED 过程通常包括两个主要步骤:候选选择和候选排序。候选选择旨在识别与命名实体提及含义相匹配的相关实体或概念。在选择候选实体之后,根据它们代表提及的正确解释的可能性对其进行排序。最终,得分最高的候选实体被选为消歧后的实体。
我们提出了一种新方法,通过结合预训练语言模型和图机器学习技术,生成生物医学实体的联合表示。图 4 展示了该方法的概览。
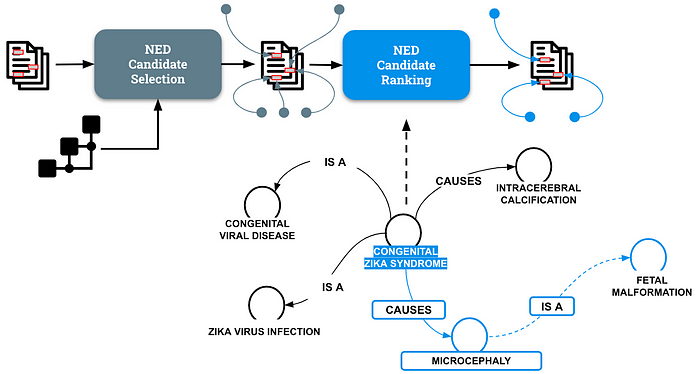
图 4 展示了该方法的核心思想:利用与“先天性 Zika 综合征”相关的图信号完成消歧阶段。该方法还考虑到周围实体具有各自的独特表示。例如,与“小头畸形”实体相关的拓扑特征与“胎儿畸形”实体相连接。
基于这一原则,图 5 展示了组成我们方法的关键步骤。
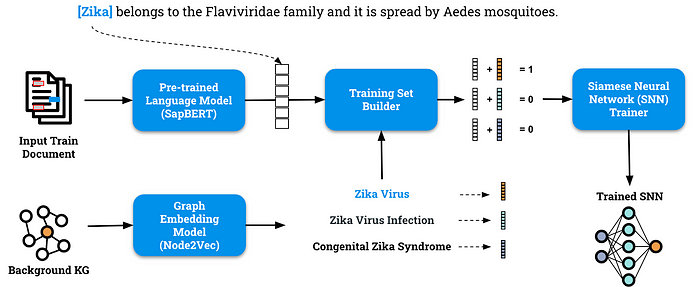
训练流程以两种不同的输入为特征:一组带注释的训练文档和用于提取基于图信号的背景知识图谱。
预训练语言模型——SapBERT
该流水线的第一部分旨在为训练数据集中每个句子的注释提及项提取上下文表示。
在我们的示例中,我们的目标是提取术语“Zika”的嵌入表示,同时考虑句子提供的上下文含义:“Zika 属于黄病毒科(Flaviviridae),通过埃及伊蚊传播。” 为了实现这一目标,我们使用了 SapBERT,这是一种基于 BERT 的模型,专为生物医学实体的表示空间自对齐而设计。
Liu, F., Shareghi, E., Meng, Z., Basaldella, M., & Collier, N. (2020). Self-alignment pretraining for biomedical entity representations. arXiv preprint arXiv:2010.11784.
图嵌入模型——Node2Vec
流水线的第二部分专注于从背景知识图谱(Knowledge Graph, KG)中提取节点的基于图的表示,这为消歧提供了信号。
为了实现这一目标,我们使用了 Node2Vec,这是一种灵活的网络探索算法,灵感来源于随机游走。从初始目标节点出发并在图中导航,Node2Vec 在广度优先和深度优先搜索策略之间取得平衡。这使得算法能够捕获每个网络节点的局部和全局邻域信息。本质上,该算法将每个节点视为单词,将每次随机游走视为句子,通过利用 Word2Vec 学习节点嵌入。
Grover, A., & Leskovec, J. (2016, August). node2vec: Scalable feature learning for networks. In Proceedings of the 22nd ACM SIGKDD international conference on Knowledge discovery and data mining (pp. 855–864).
端到端 NED 模型——孪生神经网络(Siamese Neural Networks, SNNs)
来自上述两个分支的输出随后被拼接在一起,形成一个新的特征向量,该向量既包含文本中提及实体的上下文嵌入,也包含目标实体在知识图谱中的节点嵌入。
为了优化这一联合表示,我们采用了孪生神经网络(SNN),这种架构通常包括两个具有共享权重和参数的子网络。该模型特别适合我们的场景,因为它可以处理不同长度的输入向量,例如由两个分支计算的文本嵌入和节点嵌入。
SNN 在二分类设置下进行训练,其中正例通过将文本嵌入与正确目标实体的节点嵌入拼接而成;而负例则通过将文本嵌入与候选(错误)实体的节点嵌入拼接而成。
Chicco, D. (2021). Siamese neural networks: An overview. Artificial neural networks, 73–94.
如图 6 所示,在推理阶段,SNN 模型为这种联合表示分配一个分数,用于在一组可能的候选实体中对目标实体进行消歧。
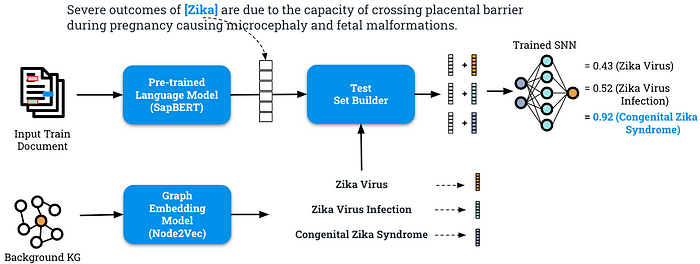
接下来让我们分析我们方法的结果。
评估与结果
我们提出的方法在多个知名的生物医学数据集(包括 MedMentions 和 BC5CDR)上进行了实证评估。对于 SNN 的训练,背景知识图谱使用了 SNOMED-CT。为了评估图嵌入的影响,我们将该方法与完全基于文本的方法以及生物医学领域广泛使用的 NED 工具 SciSpaCy 进行了比较。
评估数据集
关于注释数据集,我们使用了 MedMentions (MM) 和 BioCreative V 化学疾病关系 (BC5CDR) 数据集。
MM 是一个大规模的生物医学语料库,包含注释概念。它由 4,392 篇摘要和 352,496 个提及项组成,每个提及项都链接到一个唯一的标识符。
Mohan, S., & Li, D. (2019). Medmentions: A large biomedical corpus annotated with umls concepts. arXiv preprint arXiv:1902.09476.
BC5CDR 包含 1,500 篇文章,涵盖 4,409 个注释化学物质和 5,818 种疾病。
Li, J., Sun, Y., Johnson, R. J., Sciaky, D., Wei, C. H., Leaman, R., & Lu, Z. (2016). BioCreative V CDR task corpus: a resource for chemical disease relation extraction. Database, 2016.
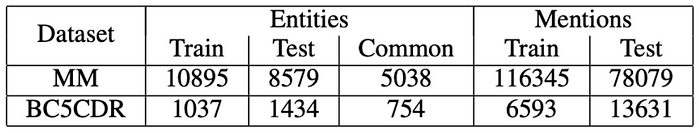
MM 数据集每篇文档的平均实体数为 44,而 BC5CDR 数据集为 14。图 7 展示了 MM 和 BC5CDR 数据集的训练、测试及重叠实体的详细信息。
在计算节点嵌入的背景知识方面,我们采用了 SNOMED-CT 本体。SNOMED-CT 是一个结构化、机器可读的医学术语集合,包括代码、术语、同义词和用于临床文档记录的解释。
Lee, D., de Keizer, N., Lau, F., & Cornet, R. (2014). Literature review of SNOMED CT use. Journal of the American Medical Informatics Association, 21(e1), e11-e19.
图 8 显示了 SNOMED-CT 在实体和关系数量方面的规模:

评估结果
为了评估所提出的架构,我们与两种不同的方法进行了比较:
1. 完全基于文本的模型,该模型利用 BERT 嵌入对知识库中的实体元数据进行编码。
2. SciSpaCy,这是生物医学领域中广泛使用的 NED 工具。
我们采用了两种不同的指标来评估我们的方法:
• Gold——正确消歧的实体数量 实体总数。
• Partial——前 5 名候选中正确消歧的实体数量 / 实体总数。
实验一——在第一个实验中,我们用 BERT 生成的文本嵌入替换了图嵌入,用于对 SNOMED 的实体元数据中的文本信号进行编码。虽然完全依赖文本特征的方法在 MM 数据集上的表现与我们的方法相当,但使用 Node2Vec 嵌入的模型在 BC5CDR 数据集上的准确性表现更优(见图 9 结果)。

这一结果尤为显著,因为 BERT 嵌入的长度为 768,而 Node2Vec 嵌入的长度仅为 128。与 MM 相比,BC5CDR 的实体类型较少,这表明如果需要消歧的数据集更为专业化,图嵌入的表现可能更优。
实验二——SciSpaCy 实体链接器通过使用 3-gram 字符串重叠搜索命名实体,并通过近似最近邻搜索将其与知识库中的概念进行比较。
为了扩展评估范围,我们不仅限于在背景知识图谱的全文索引搜索中选择排名前 50 的候选项,还研究了选择前 10 和前 100 个候选项的影响。图 10 提供了所有模型的综合比较。
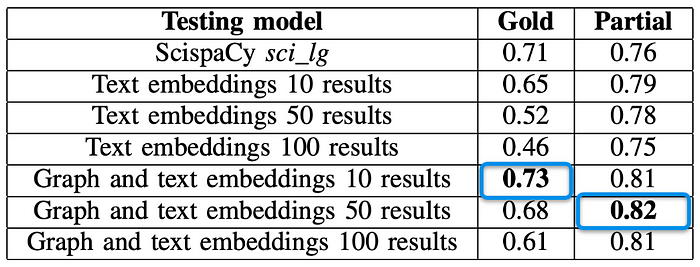
图 10 的结果显示,对于 MM 测试集,各模型在部分准确性上的最高分数相对一致。我们的方法在可靠性上略低于完全基于文本嵌入训练的模型,但差距很小,仅约 1%。
然而,对于 BC5CDR 测试集,结合了图和文本嵌入的模型在部分和 Gold 准确性上都表现最佳。尽管 SciSpaCy 模型在 Gold 准确性上表现相似,但在更宽松的评分标准下,其改进有限。
结论与未来工作
本研究探讨了一种利用生物医学知识图谱(KG)生成的图嵌入进行命名实体消歧(NED)的新方法。与广为人知的现成模型(如 SciSpaCy)或基于文本嵌入的金标准模型(如基于 BERT 的框架)相比,我们的方法表现出极具竞争力的性能。
对于包含多种实体类型的数据集(如 MedMentions),结果具有可比性。然而,对于仅包含两种实体类型的 BC5CDR 数据集,其结果甚至优于比较模型。
总之,图嵌入在 NED 中的采用有潜力推动 NLP 领域的发展,并在实体类型数量有限的领域(如流媒体音乐平台和相关推荐系统)中促进实际应用。未来的工作将重点升级模型组件,并构建当前知识图谱的扩展版本以生成图嵌入。
学习资源
若要了解更多知识图谱相关教学,你可以查看公众号的其他文章:
GPT-4o+知识图谱:法律文档检索的新革命 5种方法,让文本信息瞬间变成结构化图谱! AI + 知识图谱 = GraphRAG:为企业构建更精准的聊天机器人 利用AI大模型,将任何文本语料转化为知识图谱,可本地运行 解读 Graph RAG:从大规模文档中发现规律,找到相互关系,速度更快,信息更全面! 利用LLM构建非结构化文本的知识图谱
活水智能成立于北京,致力于通过AI教育、AI软件和社群提高知识工作者生产力。中国AIGC产业联盟理事。
活水现有AI线下工作坊等10+门课程,15+AI软件上线,多款产品在研。知识星球中拥有2600+成员,涵盖大厂程序员、公司高管、律师等各类知识工作者。在北、上、广、深、杭、重庆等地均有线下组织。
欢迎加入我们的福利群,每周都有一手信息、优惠券发放、优秀同学心得分享,还有赠书活动~
👇🏻👇🏻👇🏻







