
专业性强:术语复杂、关系网状交织(如“基因A-抑制-蛋白B-引发-疾病C”) AI 容易胡编:通用大模型常对专业领域问题“一本正经地胡说八道”
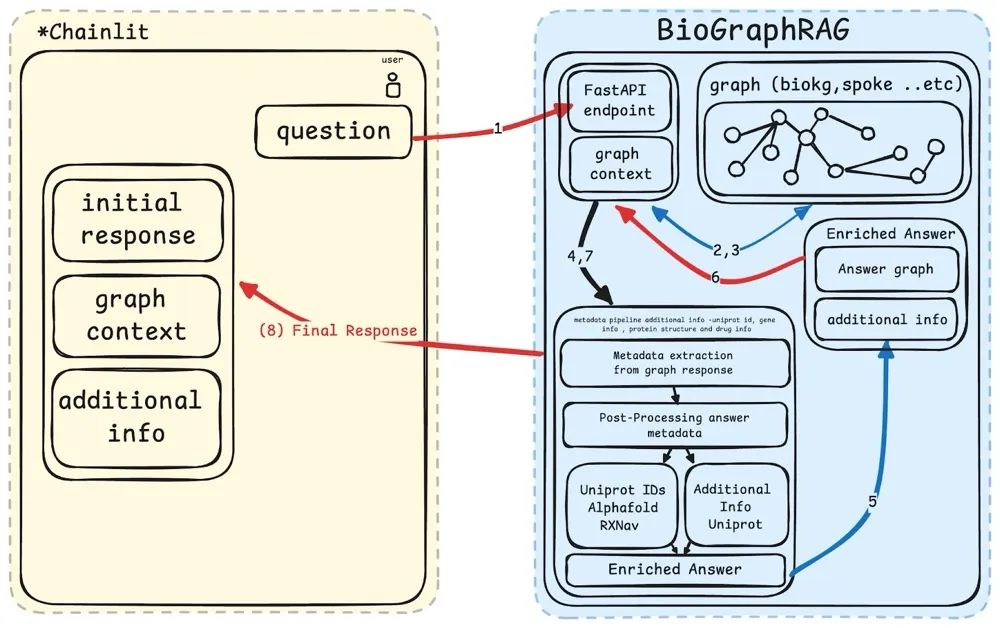
数据集:BioKG 前端交互:Chainlit 后端 API:FastAPI 图数据库:NebulaGraph 索引与检索:LlamaIndex
质量依赖性:响应的质量高度依赖于检索数据的相关性——检索效果差必然导致答案质量差。 系统复杂性:实现 RAG 涉及集成检索和生成系统,使其设计和维护更加复杂。 文档理解不全面:由于 RAG 主要依赖文档片段(chunks)来生成答案,它在理解跨多个文档和片段之间的复杂相互关系方面存在局限。 数据处理能力有限:虽然 RAG 在非结构化数据集上表现良好,但在处理图数据集和其他结构化数据集时存在困难。
答案精准且可追溯来源
能展示实体间复杂关系(药物-靶点-疾病网络)
事实准确性:在生物医学领域,事实正确且准确至关重要,误解或不准确可能带来严重后果。GraphRAG 通过利用知识图谱来提高准确性。 法规与合规性:医疗保健行业在严格的法规下运作,要求数据的透明度和可追溯性。GraphRAG 天然支持追踪检索数据的来源和关系,显著提高了可追溯性。 分散多样的数据集:生物医学领域的数据通常分散在多个来源中——如研究论文、临床试验和医学数据库。GraphRAG 的核心优势在于通过将这些分散的信息映射到统一的知识图谱上来整合它们——实现从孤立数据点到关联网络的转变,为复杂查询提供更清晰全面的理解。
灵活性:可以轻松建模复杂的互连数据。 高效性:查询关系比在传统数据库中更快。 可扩展性:可以有效地管理具有数百万节点和边的大规模图谱。
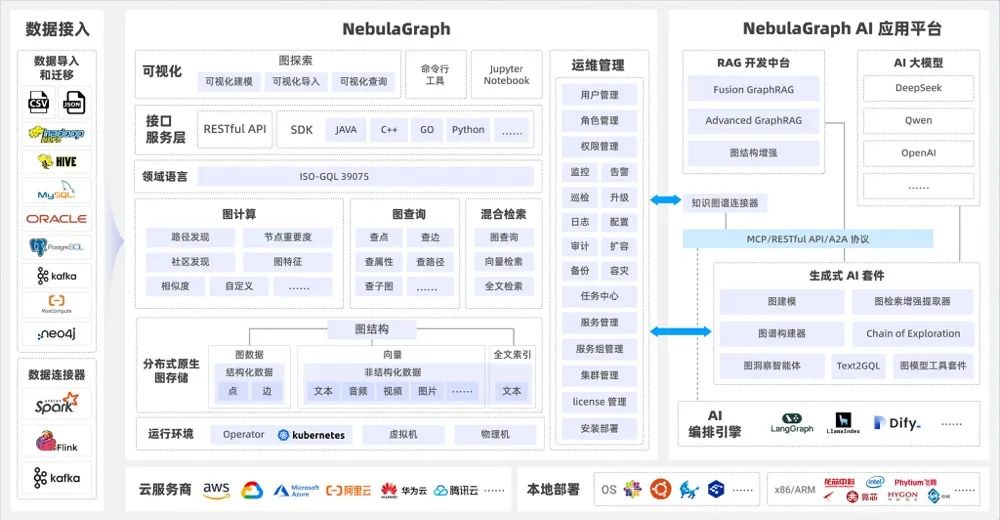
开源:NebulaGraph 遵循 Apache 2.0 协议开源,我们可以基于特定需求进行定制开发。 大规模性能:擅长处理千亿节点万亿条边的超大数据集,同时保持毫秒级查询延时。 分布式架构:NebulaGraph 的分布式特性使我们能够有效地跨多台服务器管理和查询大型数据集。 细粒度模式支持:NebulaGraph 提供灵活的模式管理,允许我们定义节点和边上适合我们知识图谱结构的特定属性。 专属查询语句:nGQL(NebulaGraph Query Language)是 NebulaGraph 为开发和运维人员设计的类 SQL 查询语言,支持灵活高效的图模式,它易于学习且功能强大,使我们能够快速查询实体之间的关系。
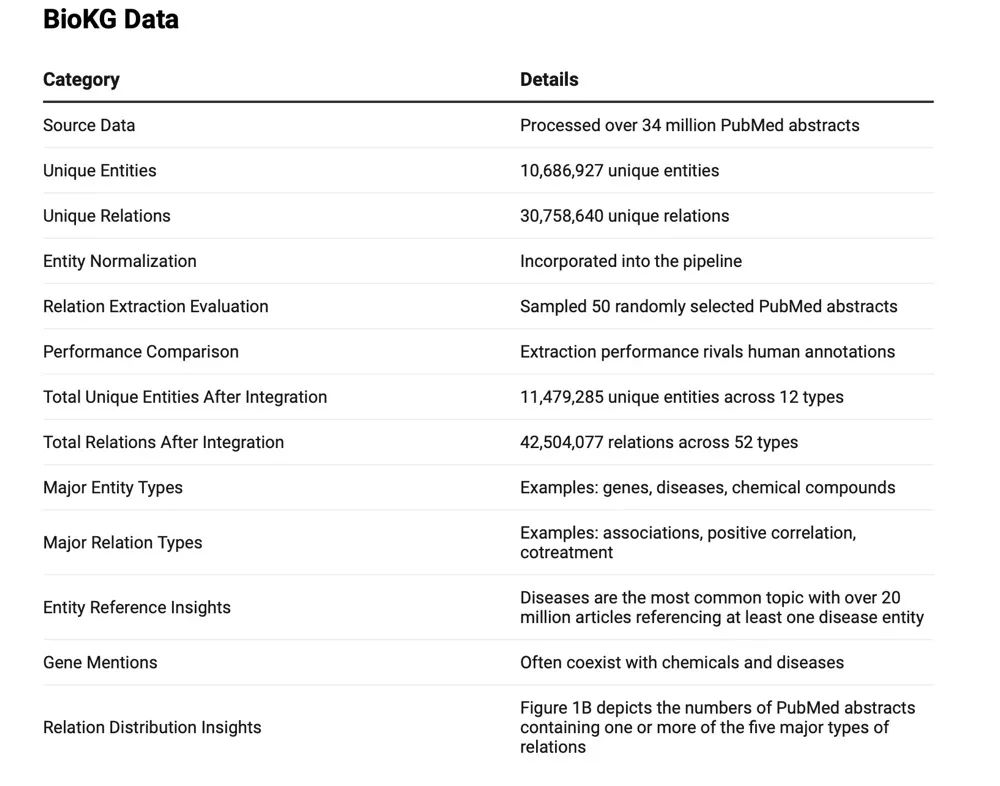
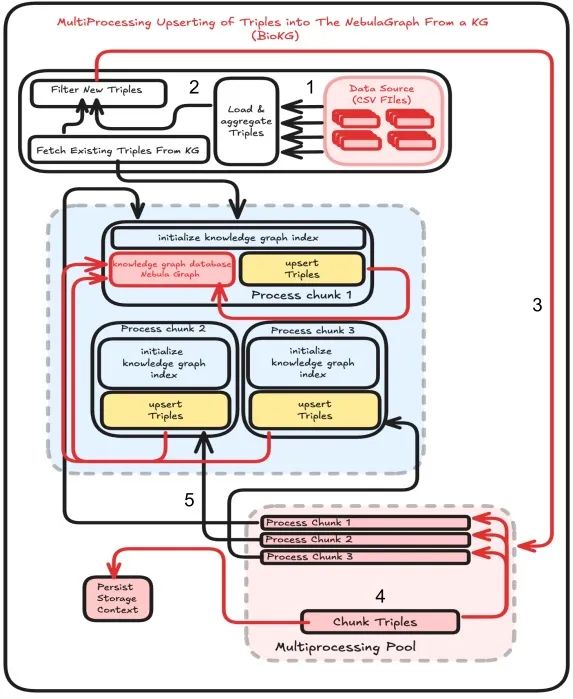
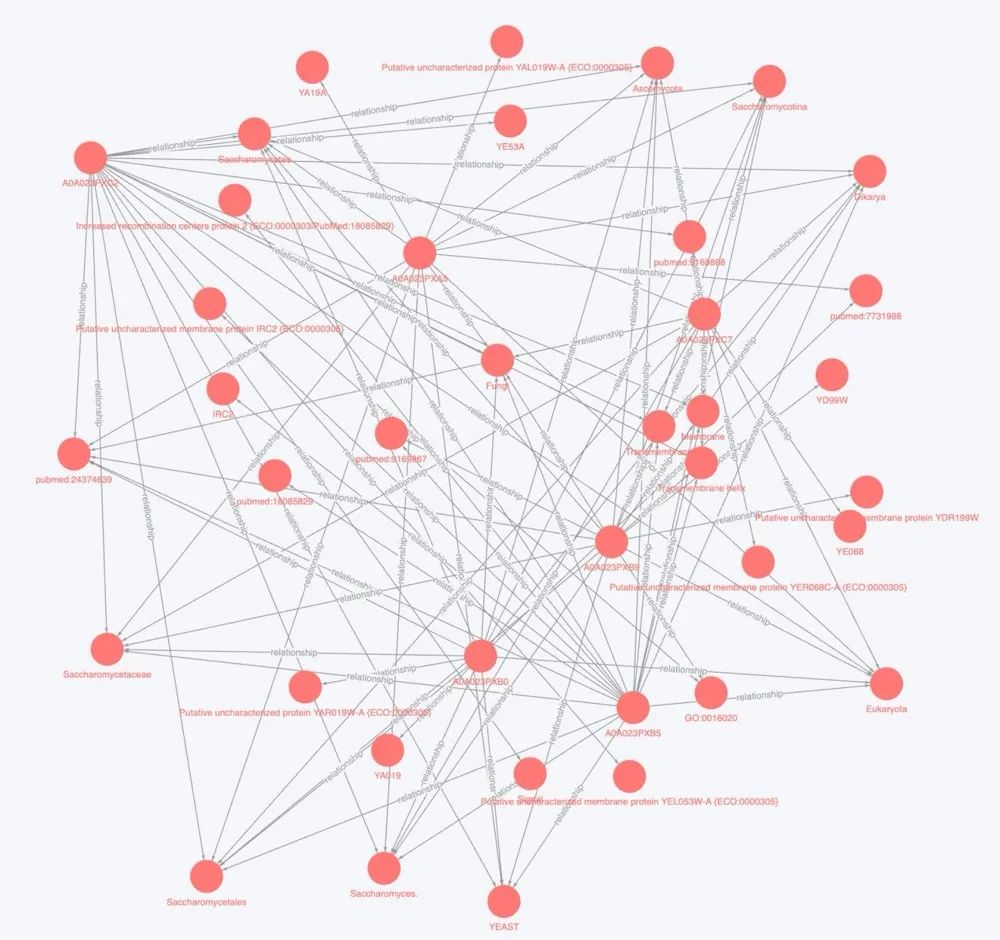
索引知识图谱:LlamaIndex 创建知识图谱中实体、关系和属性的结构化索引。这使得数据的检索更快、更精确。 检索与查询:当用户进行查询时,LlamaIndex 使用索引图检索最相关的三元组(实体-关系-实体集合),然后将其传递给 LLM 以生成答案。
用户查询: “光合作用的过程?”
MATCH (entity1:process)-[rel:relationship]->(entity2:component)
WHERE entity1.name = 'photosynthesis'
RETURN entity1, rel, entity2;``
photosynthesis -> involves -> light reaction
photosynthesis -> produces -> oxygen
photosynthesis -> uses -> chlorophyll
“光合作用涉及光反应(light reaction),使用叶绿素(chlorophyll),并产生氧气(oxygen)作为副产品。”
图谱数据集成: 从知识图谱中提取数据的能力。
图谱遍历逻辑: 导航图谱并提供相关信息的逻辑。 答案上下文映射: 能够将图谱数据转化为连贯的自然语言响应。
基于节点的交互:每个用户输入是一个节点,AI 响应由边表示。 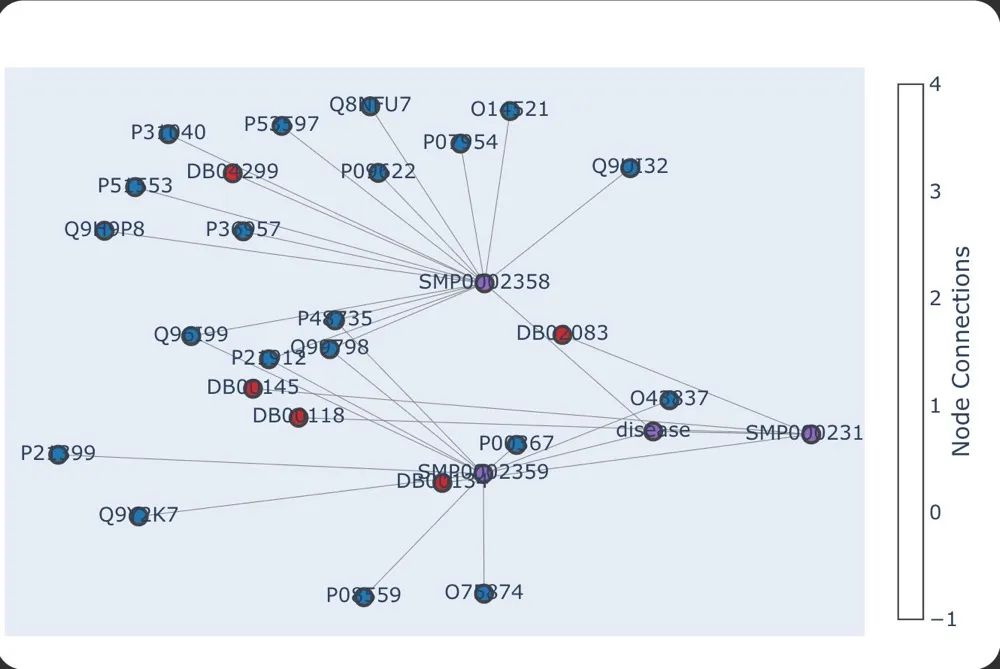
用户友好界面:可视化增强了用户对 AI 决策过程的理解。 调试与透明度:开发人员可以轻松跟踪对话演进并进行故障排除。
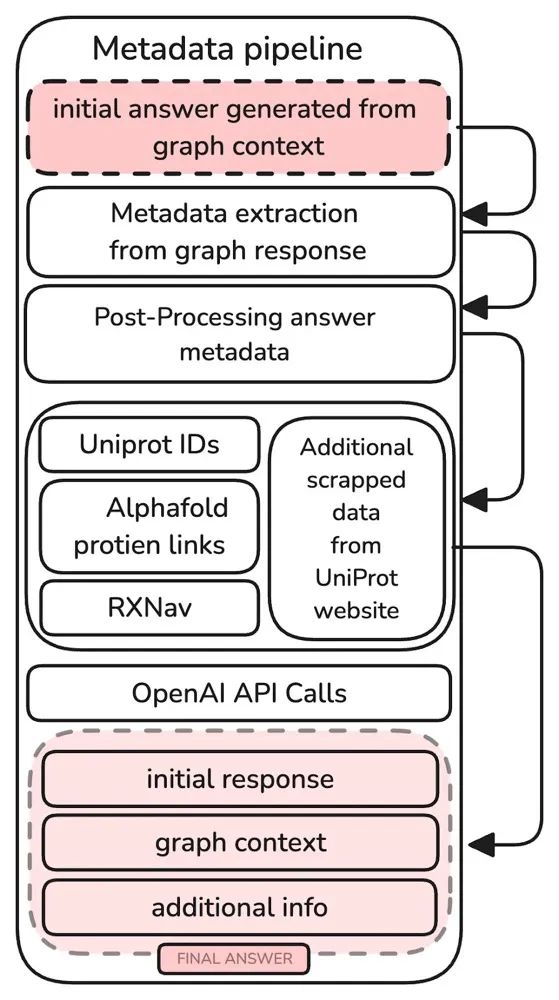
流程开始使用专门的查询引擎 kg_index_query_engine
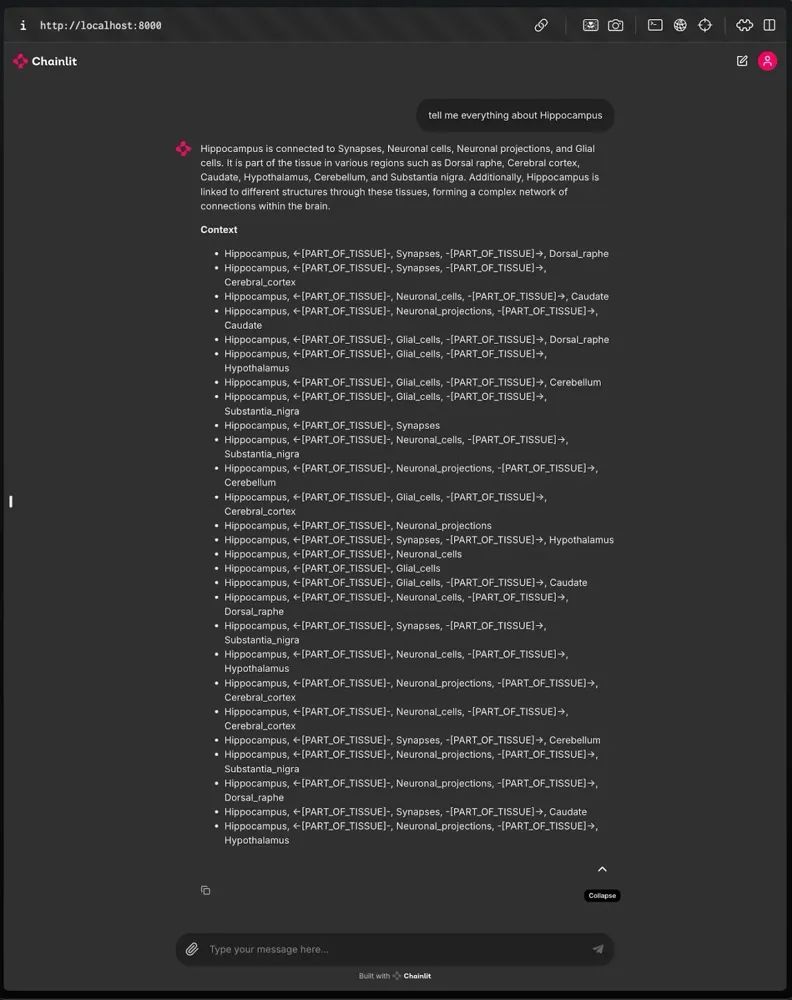
查询 BioKG.此步骤检索初始答案以及表示与问题相关的实体上下文和关系的子图。
处理来自图谱查询的响应,提取关键组件,如上下文、初始答案和子图。 这涉及解析响应并提取表示子图中关系的三元组。
通过调用查询特定元数据源的附加模块来增强初始答案。 这些包括基因相关信息、通过 AlphaFold 的蛋白质结构以及药物相关数据。 增强过程将这些附加信息与初始答案合并,以创建更全面的响应。
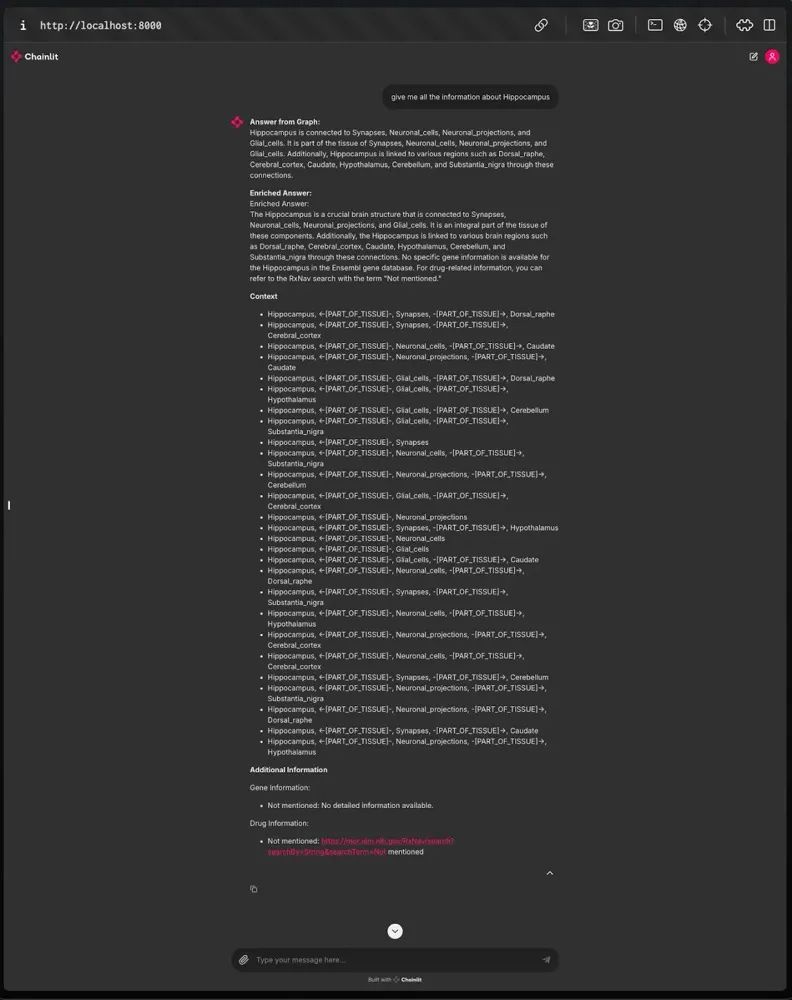
通过集成初始答案、增强内容和元数据来准备最终响应。 此响应以结构化格式包含所有相关信息,确保其全面且上下文准确。 包含相关外部资源的链接(如果可用)。
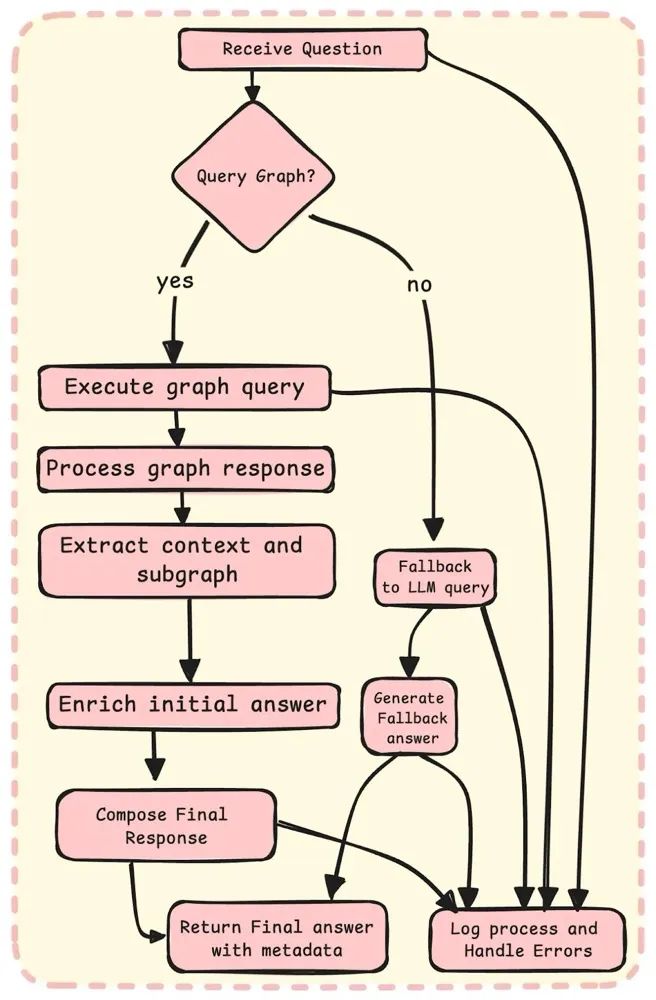
如果用户查询的信息并不在知识图谱中,BioGraphRAG 会自动回退到使用 OpenAI 驱动的 LLM 的通用查询方法。 这确保了当知识图谱覆盖不全时,也能给用户带来良好的查询体验。
在每个阶段都包含日志记录,尤其是在图谱查询和答案增强期间。 这对于调试、性能监控和确保每个组件按预期运行至关重要。 错误处理机制捕获并报告问题,而不会中断整个过程。

✦
如果你觉得 NebulaGraph 能帮到你,或者你只是单纯支持开源精神,可以在 GitHub 上为 NebulaGraph 点个 Star!每一个 Star 都是对我们的支持和鼓励✨
https://github.com/vesoft-inc/nebula
✦
✦

扫码添加
可爱星云
技术交流
资料分享
NebulaGraph 用户案例
✦
风控场景:携程|Airwallex|众安保险|中国移动|Akulaku|邦盛科技|360数科|BOSS直聘|金蝶征信|快手|青藤云安全
平台建设:博睿数据|携程|众安科技|微信|OPPO|vivo|美团|百度爱番番|携程金融|普适智能|BIGO
✦
✦



文章转载自NebulaGraph 技术社区,如果涉嫌侵权,请发送邮件至:contact@modb.pro进行举报,并提供相关证据,一经查实,墨天轮将立刻删除相关内容。






