本文为DataWhale学习笔记
一、概述
1、什么是异常检测
异常检测(Outlier Detection),顾名思义,是识别与正常数据不同的数据,与预期行为差异大的数据。识别如信用卡欺诈,工业生产异常,网络流里的异常(网络侵入)等问题,针对的是少数的事件。
1.1 异常的类别
点异常: 少数个体实例是异常的,大多数个体实例是正常的,例如正常人与病人的健康指标;
上下文异常: 在特定情境下个体实例是异常的,在其他情境下都是正常的,例如在特定时间下的温度突然上升或下降,在特定场景中的快速信用卡交易;
群体异常: 在群体集合中的个体实例出现异常的情况,而该个体实例自身可能不是异常,例如社交网络中虚假账号形成的集合作为群体异常子集,但子集中的个体节点可能与真实账号一样正常。
1.2 异常检测任务分类
有监督: 训练集的正例和反例均有标签
无监督: 训练集⽆标签
半监督: 在训练集中只有单一类别(正常实例)的实例,没有异常实例参与训练
1.3 异常检测场景
故障检测 物联网异常检测 欺诈检测 工业异常检测 时间序列异常检测 视频异常检测 日志异常检测 医疗日常检测 网络入侵检测
2、异常检测常用方法
2.1 传统方法
2.1.1 基于统计学的方法
统计学的方法对数据的正常性做出假定。它们假设正常的数据对象由一个统计模型产生,而不遵守该模型的数据是异常点。 统计学方法的有效性高度依赖于给定数据所做的统计模型假定是否成立。
异常检测的统计学方法的一般思想是:学习一个拟合给定数据集的生成模型,然后识别该模型低概率区域中的对象,把它们作为异常点。
即利用统计学方法建立一个模型,然后考虑对象有多大可能符合该模型。
假定输入数据集为,数据集中的样本服从正态分布,即,我们可以根据样本求出参数和。
2.1.2 线性模型
典型的如PCA方法,Principle Component Analysis是主成分分析,简称PCA。它的应用场景是对数据集进行降维。降维后的数据能够最大程度地保留原始数据的特征(以数据协方差为衡量标准)。PCA的原理是通过构造一个新的特征空间,把原数据映射到这个新的低维空间里。PCA可以提高数据的计算性能,并且缓解"高维灾难"。
2.1.3 基于相似度的方法
这类算法适用于数据点的聚集程度高、离群点较少的情况。同时,因为相似度算法通常需要对每一个数据分别进行相应计算,所以这类算法通常计算量大,不太适用于数据量大、维度高的数据。
基于相似度的检测方法大致可以分为三类:
基于集群(簇)的检测,如 DBSCAN等聚类算法。
聚类算法是将数据点划分为一个个相对密集的“簇",而那些不能被归为某个簇的点,则被视作离群点。这类算法对簇个数的选择高度敏感,数量选择不当可能造成较多正常值被划为离群点或成小簇的离群点被归为正常。因此对于每一个数据集需要设置特定的参数,才可以保证聚类的效果,在数据集之间的通用性较差。聚类的主要目的通常是为了寻找成簇的数据,而将异常值和噪声一同作为无价值的数据而忽略或丢弃,在专门的异常点检测中使用较少。
聚类算法的优缺点
能够较好发现小簇的异常; 通常用于簇的发现,而对异常值采取丢弃处理,对异常值的处理不够友好; 产生的离群点集和它们的得分可能非常依赖所用的簇的个数和数据中离群点的存在性; 聚类算法产生的簇的质量对该算法产生的离群点的质量影响非常大。
基于距离的度量,如k近邻算法。
k近邻算法的基本思路是对每一个点,计算其与最近k个相邻点的距离,通过距离的大小来判断它是否为离群点。在这里,离群距离大小对k的取值高度敏感。如果k太小(例如1),则少量的邻近离群点可能导致较低的离群点得分;如果k太大,则点数少于k的簇中所有的对象可能都成了离群点。为了使模型更加稳定,距离值的计算通常使用k个最近邻的平均距离。
k近邻算法的优缺点
简单 基于邻近度的方法需要O(m2)时间,大数据集不适用; 对参数的选择敏感; 不能处理具有不同密度区域的数据集,因为它使用全局阈值,不能考虑这种密度的变化
基于密度的度量,如LOF(局部离群因子)算法。
局部离群因子(LOF)算法与k近邻类似,不同的是它以相对于其邻居的局部密度偏差而不是距离来进行度量。它将相邻点之间的距离进一步转化为邻域",从而得到邻域中点的数量(即密度),认为密度远低于其邻居的样本为异常值。
LOF(局部离群因子)算法的优缺点:
给出了对离群度的定量度量; 能够很好地处理不同密度区域的数据; 对参数的选择敏感
2.2 集成方法
集成是提高数据挖掘算法精度的常用方法。集成方法将多个算法或多个基检测器的输出结合起来。其基本思想是一些算法在某些子集上表现很好,一些算法在其他子集上表现很好,然后集成起来使得输岀更加鲁棒。集成方法与基于子空间方法有着天然的相似性,子空间与不同的点集相关,而集成方法使用基检测器来探索不同维度的子集,将这些基学习器集合起来。
常用的集成方法有 Feature bagging,孤立森林等。
feature bagging
与 bagging法类似,只是对象是 feature。
孤立森林
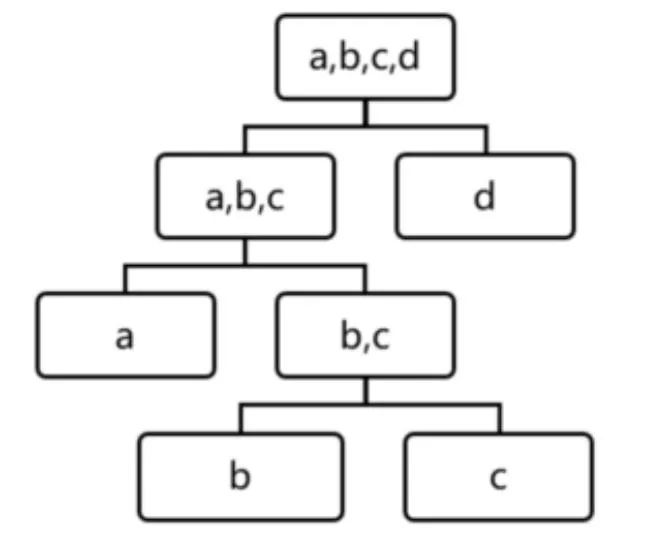
孤立森林假设我们用一个随机超平面来切割数据空间,切一次可以生成两个子空间。然后我们继续用随机超平面来切割每个子空间并循环,直到每个子空间只有一个数据点为止。直观上来讲,那些具有高密度的簇需要被切很多次才会将其分离,而那些低密度的点很快就被单独分配到一个子空间了。孤立森林认为这些很快被孤立的点就是异常点。
用四个样本做简单直观的理解,d是最早被孤立出来的,所以d最有可能是异常。
2.3 机器学习
在有标签的情况下,可以使用树模型( gbdt, xgboost等)进行分类,缺点是异常检测场景下数据标签是不均衡的,但是利用机器学习算法的好处是可以构造不同特征。
3、异常检测常用开源库
Scikit-Learn:
Scikit-Learn是一个Python语言的开源机器学习库。它具有各种分类,回归和聚类算法。也包含了一些异常检测算法,例如LOF和孤立森林。
PyOD:
Python Outlier Detection(PyoD)是当下最流行的 Python异常检测工具库,其主要亮点包括:
包括近20种常见的异常检测算法,比如经典的LOF/LOCI/ABOD以及最新的深度学习如对抗生成模型(GAN)和集成异常检测( outlier ensemble)
支持不同版本的Python:包括2.7和3.5+;支持多种操作系统:windows, macos和 Linux
简单易用且一致的API,只需要几行代码就可以完成异常检测,方便评估大量算法
使用JIT和并行化( parallelization)进行优化,加速算法运行及扩展性( scalability),可以处理大量数据
——https://zhuanlan.zhihu.com/p/5831352
4、练习
4.1 Scikit-Learn
Sklearn提供了一些机器学习方法,可用于奇异(Novelty)点或异常(Outlier)点检测,包括OneClassSVM、Isolation Forest、Local Outlier Factor (LOF) 等。其中OneClassSVM可用于Novelty Detection,而后两者可用于Outlier Detection。
novelty detection:当训练数据中没有离群点,我们的目标是用训练好的模型去检测另外新发现的样本。新奇检测的前提是已知训练数据集是“纯净”的,未被真正的“噪音”数据或真实的“离群点”污染,然后针对这些数据训练完成之后再对新的数据进行训练以寻找异常数据。 outlier detection:当训练数据中包含离群点,模型训练时要匹配训练数据的中心样本,忽视训练样本中的其它异常点。离群点检测的训练数据集则包含“离群点”数据,对这些数据训练完成之后再在新的数据集中寻找异常数据。
4.1.1 OneClassSVM
OneClass SVM 是一個非监督学习的算法,顾名思义训练数据只有一个分类。透过这些正常样本的特征取学习一个决策边界,再透过这个边界去判别新的数据是否与训练数据类似。超出边界即视为异常。
OneClass SVM的训练集不应该掺杂异常点,因为模型可能会去匹配这些异常点。但在数据维度很高,或者对相关数据分布没有任何假设的情况下,OneClassSVM也可以作为一种很好的outlier detection方法。在one-class classification中,仅仅只有一类的信息是可以用于训练,其他类别的(总称outlier)信息是缺失的,也就是区分两个类别的边界线是通过仅有的一类数据的信息学习得到的。
那么没有类别标签,我们如何寻找划分的超平面以及寻找支持向量呢?One Class SVM这个问题的解决思路有很多。这里只讲解一种特别的思路SVDD, 对于SVDD来说,我们期望所有不是异常的样本都是正类别,同时它采用一个超球体而不是一个超平面来做划分,该算法在特征空间中获得数据周围的球形边界,期望最小化这个超球体的体积,从而最小化异常点数据的影响。
假设产生的超球体参数为中心和对应的超球体半径 ,超球体体积 被最小化,中心是支持向量的线性组合;跟传统SVM方法相似,可以要求所有训练数据点到中心的距离严格小于,但同时构造一个惩罚系数为的松弛变量,优化问题如下所示:
import numpy as np
import matplotlib.pyplot as plt
import matplotlib.font_manager
from sklearn import svm
xx, yy = np.meshgrid(np.linspace(-5, 5, 500), np.linspace(-5, 5, 500))
# Generate train data
X = 0.3 * np.random.randn(100, 2)
X_train = np.r_[X + 2, X - 2]
# Generate some regular novel observations
X = 0.3 * np.random.randn(20, 2)
X_test = np.r_[X + 2, X - 2]
# Generate some abnormal novel observations
X_outliers = np.random.uniform(low=-4, high=4, size=(20, 2))
# fit the model
clf = svm.OneClassSVM(nu=0.1, kernel="rbf", gamma=0.1)
clf.fit(X_train)
y_pred_train = clf.predict(X_train)
y_pred_test = clf.predict(X_test)
y_pred_outliers = clf.predict(X_outliers)
n_error_train = y_pred_train[y_pred_train == -1].size
n_error_test = y_pred_test[y_pred_test == -1].size
n_error_outliers = y_pred_outliers[y_pred_outliers == 1].size
# plot the line, the points, and the nearest vectors to the plane
Z = clf.decision_function(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
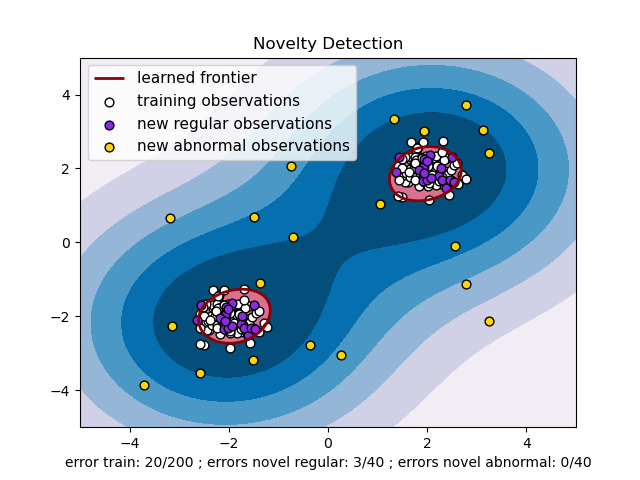
plt.title("Novelty Detection")
plt.contourf(xx, yy, Z, levels=np.linspace(Z.min(), 0, 7), cmap=plt.cm.PuBu) # 绘制异常样本的区域
a = plt.contour(xx, yy, Z, levels=[0], linewidths=2, colors='darkred') # 绘制正常样本和异常样本的边界
plt.contourf(xx, yy, Z, levels=[0, Z.max()], colors='palevioletred') # 绘制正常样本的区域
s = 40
b1 = plt.scatter(X_train[:, 0], X_train[:, 1], c='white', s=s, edgecolors='k')
b2 = plt.scatter(X_test[:, 0], X_test[:, 1], c='blueviolet', s=s,
edgecolors='k')
c = plt.scatter(X_outliers[:, 0], X_outliers[:, 1], c='gold', s=s,
edgecolors='k')
plt.axis('tight')
plt.xlim((-5, 5))
plt.ylim((-5, 5))
plt.legend([a.collections[0], b1, b2, c],
["learned frontier", "training observations",
"new regular observations", "new abnormal observations"],
loc="upper left",
prop=matplotlib.font_manager.FontProperties(size=11))
plt.xlabel(
"error train: %d/200 ; errors novel regular: %d/40 ; "
"errors novel abnormal: %d/40"
% (n_error_train, n_error_test, n_error_outliers))
plt.show()
4.1.2 Isolation Forest
Isolation Forest(以下简称iForest)主要是利用集成学习的思路来做异常点检测,目前几乎成为异常点检测算法的首选项。iForest适用于连续数据(Continuous numerical data)的异常检测,将异常定义为“容易被孤立的离群点(more likely to be separated)可以理解为分布稀疏且离密度高的群体较远的点。用统计学来解释,在数据空间里面,分布稀疏的区域表示数据发生在此区域的概率很低,因此可以认为落在这些区域里的数据是异常的。通常用于网络安全中的攻击检测和流量异常等分析,金融机构则用于挖掘出欺诈行为。对于找出的异常数据,然后要么直接清除异常数据,如数据清理中的去噪数据,要么深入分析异常数据,比如分析攻击,欺诈的行为特征。
算法本身并不复杂,主要包括第一步训练构建随机森林对应的多颗决策树,这些决策树一般叫,第二步计算需要检测的数据点最终落在任意第颗的层数。然后我们可以得出在每棵树的高度平均值。第三步根据判断是否是异常点。
第一步构建决策树的过程,方法和普通的随机森林不同。首先采样决策树的训练样本时,普通的随机森林要采样的样本个数等于训练集个数。但是iForest不需要采样这么多,一般来说,采样个数要远远小于训练集个数。原因是我们的目的是异常点检测,只需要部分的样本我们一般就可以将异常点区别出来了。另外就是在做决策树分裂决策时,由于我们没有标记输出,所以没法计算基尼系数或者和方差之类的划分标准。这里我们使用的是随机选择划分特征,然后在基于这个特征再随机选择划分阈值,进行决策树的分裂。直到树的深度达到限定阈值或者样本数只剩一个。 第二步计算要检测的样本点在每棵树的高度平均值。首先需要遍历每一颗,得到检测的数据点最终落在任意第颗的数层数。这个代表的是树的深度,也就是离根节点越近,则越小,越靠近底层,则越大 第三步是据判断是否是异常点。我们一般用下面的公式计算的异常概率分值:,的取值范围是,取值越接近于1,则是异常点的概率也越大。其中,为样本个数。的表达式为:,是欧拉常数。
从表示式可以看出,如果高度, 则,即是异常点的概率是100%,如果高度, 则,即不可能是异常点。如果高度, 则,即是异常点的概率是50%,一般我们可以设置的一个阈值然后去调参,这样大于阈值的才认为是异常点。
import numpy as np
import matplotlib.pyplot as plt
from sklearn.ensemble import IsolationForest
rng = np.random.RandomState(42)
# Generate train data
X = 0.3 * rng.randn(100, 2)
X_train = np.r_[X + 2, X - 2]
# Generate some regular novel observations
X = 0.3 * rng.randn(20, 2)
X_test = np.r_[X + 2, X - 2]
# Generate some abnormal novel observations
X_outliers = rng.uniform(low=-4, high=4, size=(20, 2))
# fit the model
clf = IsolationForest(max_samples=100, random_state=rng)
clf.fit(X_train)
y_pred_train = clf.predict(X_train)
y_pred_test = clf.predict(X_test)
y_pred_outliers = clf.predict(X_outliers)
# plot the line, the samples, and the nearest vectors to the plane
xx, yy = np.meshgrid(np.linspace(-5, 5, 50), np.linspace(-5, 5, 50))
Z = clf.decision_function(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
plt.title("IsolationForest")
plt.contourf(xx, yy, Z, cmap=plt.cm.Blues_r)
b1 = plt.scatter(X_train[:, 0], X_train[:, 1], c='white',
s=20, edgecolor='k')
b2 = plt.scatter(X_test[:, 0], X_test[:, 1], c='green',
s=20, edgecolor='k')
c = plt.scatter(X_outliers[:, 0], X_outliers[:, 1], c='red',
s=20, edgecolor='k')
plt.axis('tight')
plt.xlim((-5, 5))
plt.ylim((-5, 5))
plt.legend([b1, b2, c],
["training observations",
"new regular observations", "new abnormal observations"],
loc="upper left")
plt.show()
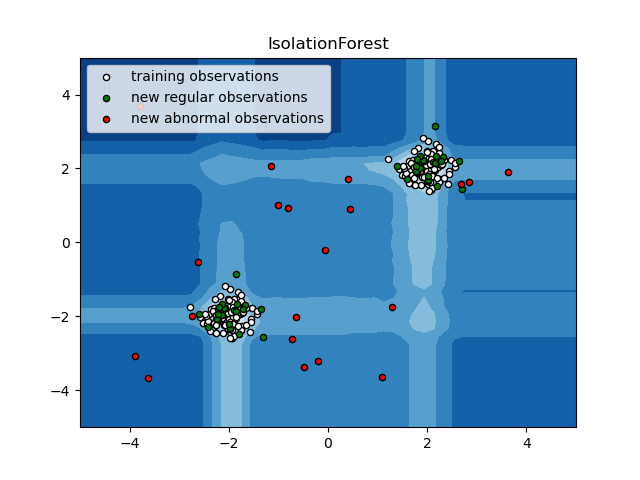
iForest目前是异常点检测最常用的算法之一,它的优点非常突出,它具有线性时间复杂度。
iForest具有线性时间复杂度,因为是ensemble的方法,所以可以用在含有海量数据的数据集上面,通常树的数量越多,算法越稳定。由于每棵树都是相互独立生成的,因此可以部署在大规模分布式系统上来加速运算。 iForest不适用于特别高维的数据。由于每次切数据空间都是随机选取一个维度,建完树后仍然有大量的维度信息没有被使用,导致算法可靠性降低。高维空间还可能存在大量噪音维度或者无关维度(irrelevant attributes),影响树的构建。对这类数据,建议使用子空间异常检测(Subspace Anomaly Detection)技术。此外,切割平面默认是axis-parallel的,也可以随机生成各种角度的切割平面。 IForest仅对Global Anomaly敏感,即全局稀疏点敏感,不擅长处理局部的相对稀疏点(Local Anomaly)。 iForest推动了重心估计(Mass Estimation)理论,目前在分类聚类和异常检测中都取得显著效果
4.1.3 Local Outlier Factor
Local Outlier Factor(LOF)基于密度的经典算法(Breuning et. al. 2000)。在 LOF 之前的异常检测算法大多是基于统计方法的,或者是借用了一些聚类算法用于异常点的识别(比如 ,DBSCAN,OPTICS)。但是,基于统计的异常检测算法通常需要假设数据服从特定的概率分布,这个假设往往是不成立的。而聚类的方法通常只能给出 的判断(即:是不是异常点),不能量化每个数据点的异常程度。相比较而言,基于密度的LOF算法要更简单、直观。它不需要对数据的分布做太多要求,还能量化每个数据点的异常程度(outlierness)。
LOF算法是一种无监督的异常检测方法,它计算给定数据点相对于其邻居的局部密度偏差。每个样本的异常分数称为局部异常因子。异常分数是局部的,取决于样本相对于周围邻域的隔离程度。确切地说,局部性由k近邻给出,并使用距离估计局部密度。通过将样本的局部密度与其邻居的局部密度进行比较,可以识别密度明显低于其邻居的样本,,这些样本就被当做是异常样本点。
算法原理如下:
计算:计算点的第距离,也就距离样本点第远的点的距离,不包括; 计算:计算点的第邻域距离,就是的第距离以内的所有点,包括第距离; 计算:可达距离,若小于第距离,则可达距离为第距离,若大于第距离,则可达距离为真实距离,公式如下(为到的距离):。点到点的第可达距离,至少是点的第距离,或者为与间的真实距离。 计算:局部可达密度。。表示点的第邻域内点到点的平均可达距离的倒数。 计算:局部离群因子。 。表示点的邻域点的局部可达密度与点p的局部可达密度之比的平均数。
import numpy as np
import matplotlib.pyplot as plt
from sklearn.neighbors import LocalOutlierFactor
from scipy import stats
# 构造训练样本
n_samples = 200 # 样本总数
outliers_fraction = 0.25 # 异常样本比例
n_inliers = int((1. - outliers_fraction) * n_samples)
n_outliers = int(outliers_fraction * n_samples)
rng = np.random.RandomState(42)
X = 0.3 * rng.randn(n_inliers // 2, 2)
X_train = np.r_[X + 2, X - 2] # 正常样本
X_train = np.r_[X_train, np.random.uniform(low=-6, high=6, size=(n_outliers, 2))] # 正常样本加上异常样本
# fit the model
clf = LocalOutlierFactor(n_neighbors=35, contamination=outliers_fraction)
y_pred = clf.fit_predict(X_train)
scores_pred = clf.negative_outlier_factor_
threshold = stats.scoreatpercentile(scores_pred, 100 * outliers_fraction) # 根据异常样本比例,得到阈值,用于绘图
# plot the level sets of the decision function
xx, yy = np.meshgrid(np.linspace(-7, 7, 50), np.linspace(-7, 7, 50))
Z = clf._decision_function(np.c_[xx.ravel(), yy.ravel()]) # 类似scores_pred的值,值越小越有可能是异常点
Z = Z.reshape(xx.shape)
plt.title("Local Outlier Factor (LOF)")
# plt.contourf(xx, yy, Z, cmap=plt.cm.Blues_r)
plt.contourf(xx, yy, Z, levels=np.linspace(Z.min(), threshold, 7), cmap=plt.cm.Blues_r) # 绘制异常点区域,值从最小的到阈值的那部分
a = plt.contour(xx, yy, Z, levels=[threshold], linewidths=2, colors='red') # 绘制异常点区域和正常点区域的边界
plt.contourf(xx, yy, Z, levels=[threshold, Z.max()], colors='palevioletred') # 绘制正常点区域,值从阈值到最大的那部分
b = plt.scatter(X_train[:-n_outliers, 0], X_train[:-n_outliers, 1], c='white',
s=20, edgecolor='k')
c = plt.scatter(X_train[-n_outliers:, 0], X_train[-n_outliers:, 1], c='black',
s=20, edgecolor='k')
plt.axis('tight')
plt.xlim((-7, 7))
plt.ylim((-7, 7))
plt.legend([a.collections[0], b, c],
['learned decision function', 'true inliers', 'true outliers'],
loc="upper left")
plt.show()
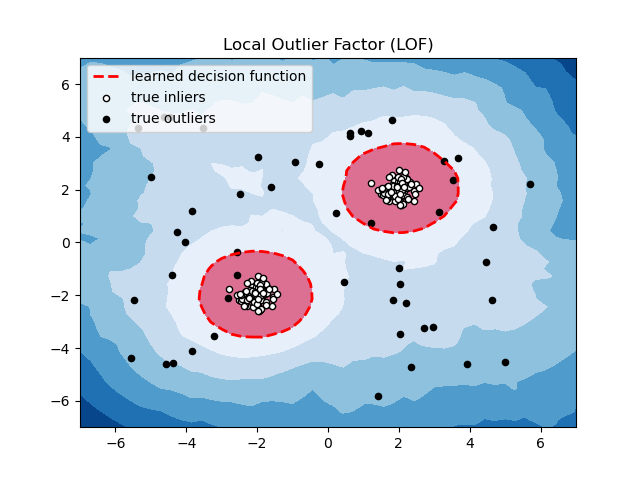
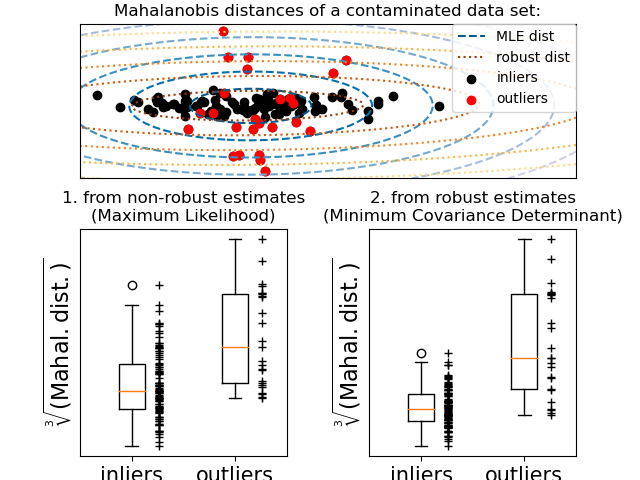
4.1.4 Fitting an Elliptic Envelope
实现异常值检测的一种常见方式是假设内围数据来自已知分布(例如,数据服从高斯分布)。从这个假设来看,我们通常试图定义数据的 “形状”,并且可以将异常观测定义为足够远离拟合形状的观测。
scikit-learn 提供了covariance.EllipticEnvelope对象,它能拟合出数据的稳健协方差估计,从而为中心数据点拟合出一个椭圆,忽略中心模式之外的点。例如,假设内围数据服从高斯分布,它将稳健地(即不受异常值的影响)估计内围位置和协方差。从该估计得到的马氏距离用于得出异常度量。该策略如下图所示:
import numpy as np
import matplotlib.pyplot as plt
from sklearn.covariance import EmpiricalCovariance, MinCovDet
n_samples = 125
n_outliers = 25
n_features = 2
# generate data
gen_cov = np.eye(n_features)
gen_cov[0, 0] = 2.
X = np.dot(np.random.randn(n_samples, n_features), gen_cov)
# add some outliers
outliers_cov = np.eye(n_features)
outliers_cov[np.arange(1, n_features), np.arange(1, n_features)] = 7.
X[-n_outliers:] = np.dot(np.random.randn(n_outliers, n_features), outliers_cov)
# fit a Minimum Covariance Determinant (MCD) robust estimator to data
robust_cov = MinCovDet().fit(X)
# compare estimators learnt from the full data set with true parameters
emp_cov = EmpiricalCovariance().fit(X)
# Display results
fig = plt.figure()
plt.subplots_adjust(hspace=-.1, wspace=.4, top=.95, bottom=.05)
# Show data set
subfig1 = plt.subplot(3, 1, 1)
inlier_plot = subfig1.scatter(X[:, 0], X[:, 1],
color='black', label='inliers')
outlier_plot = subfig1.scatter(X[:, 0][-n_outliers:], X[:, 1][-n_outliers:],
color='red', label='outliers')
subfig1.set_xlim(subfig1.get_xlim()[0], 11.)
subfig1.set_title("Mahalanobis distances of a contaminated data set:")
# Show contours of the distance functions
xx, yy = np.meshgrid(np.linspace(plt.xlim()[0], plt.xlim()[1], 100),
np.linspace(plt.ylim()[0], plt.ylim()[1], 100))
zz = np.c_[xx.ravel(), yy.ravel()]
mahal_emp_cov = emp_cov.mahalanobis(zz)
mahal_emp_cov = mahal_emp_cov.reshape(xx.shape)
emp_cov_contour = subfig1.contour(xx, yy, np.sqrt(mahal_emp_cov),
cmap=plt.cm.PuBu_r,
linestyles='dashed')
mahal_robust_cov = robust_cov.mahalanobis(zz)
mahal_robust_cov = mahal_robust_cov.reshape(xx.shape)
robust_contour = subfig1.contour(xx, yy, np.sqrt(mahal_robust_cov),
cmap=plt.cm.YlOrBr_r, linestyles='dotted')
subfig1.legend([emp_cov_contour.collections[1], robust_contour.collections[1],
inlier_plot, outlier_plot],
['MLE dist', 'robust dist', 'inliers', 'outliers'],
loc="upper right", borderaxespad=0)
plt.xticks(())
plt.yticks(())
# Plot the scores for each point
emp_mahal = emp_cov.mahalanobis(X - np.mean(X, 0)) ** (0.33)
subfig2 = plt.subplot(2, 2, 3)
subfig2.boxplot([emp_mahal[:-n_outliers], emp_mahal[-n_outliers:]], widths=.25)
subfig2.plot(np.full(n_samples - n_outliers, 1.26),
emp_mahal[:-n_outliers], '+k', markeredgewidth=1)
subfig2.plot(np.full(n_outliers, 2.26),
emp_mahal[-n_outliers:], '+k', markeredgewidth=1)
subfig2.axes.set_xticklabels(('inliers', 'outliers'), size=15)
subfig2.set_ylabel(r"$\sqrt[3]{\rm{(Mahal. dist.)}}$", size=16)
subfig2.set_title("1. from non-robust estimates\n(Maximum Likelihood)")
plt.yticks(())
robust_mahal = robust_cov.mahalanobis(X - robust_cov.location_) ** (0.33)
subfig3 = plt.subplot(2, 2, 4)
subfig3.boxplot([robust_mahal[:-n_outliers], robust_mahal[-n_outliers:]],
widths=.25)
subfig3.plot(np.full(n_samples - n_outliers, 1.26),
robust_mahal[:-n_outliers], '+k', markeredgewidth=1)
subfig3.plot(np.full(n_outliers, 2.26),
robust_mahal[-n_outliers:], '+k', markeredgewidth=1)
subfig3.axes.set_xticklabels(('inliers', 'outliers'), size=15)
subfig3.set_ylabel(r"$\sqrt[3]{\rm{(Mahal. dist.)}}$", size=16)
subfig3.set_title("2. from robust estimates\n(Minimum Covariance Determinant)")
plt.yticks(())
plt.show()
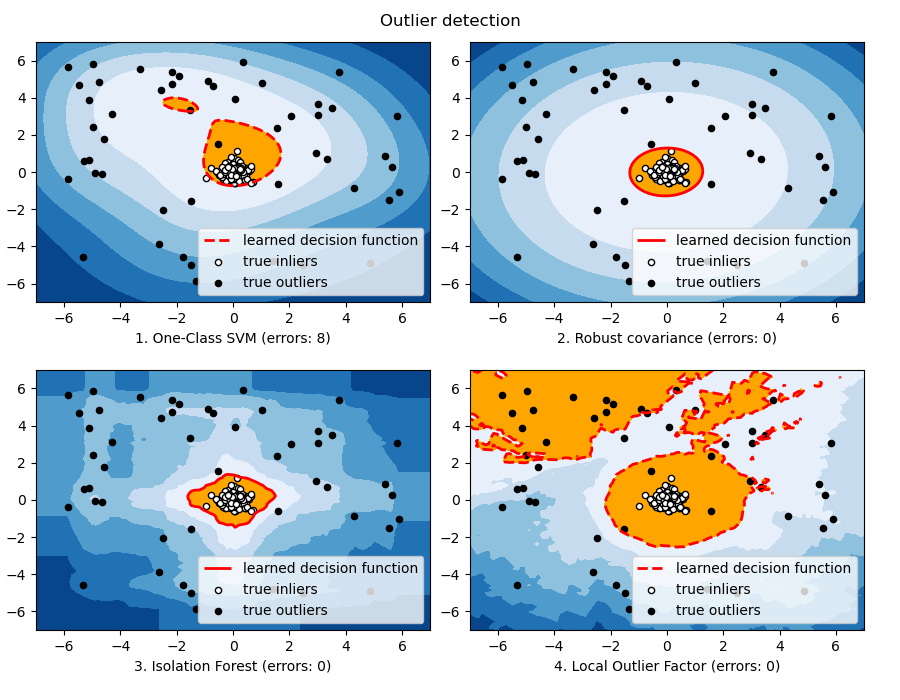
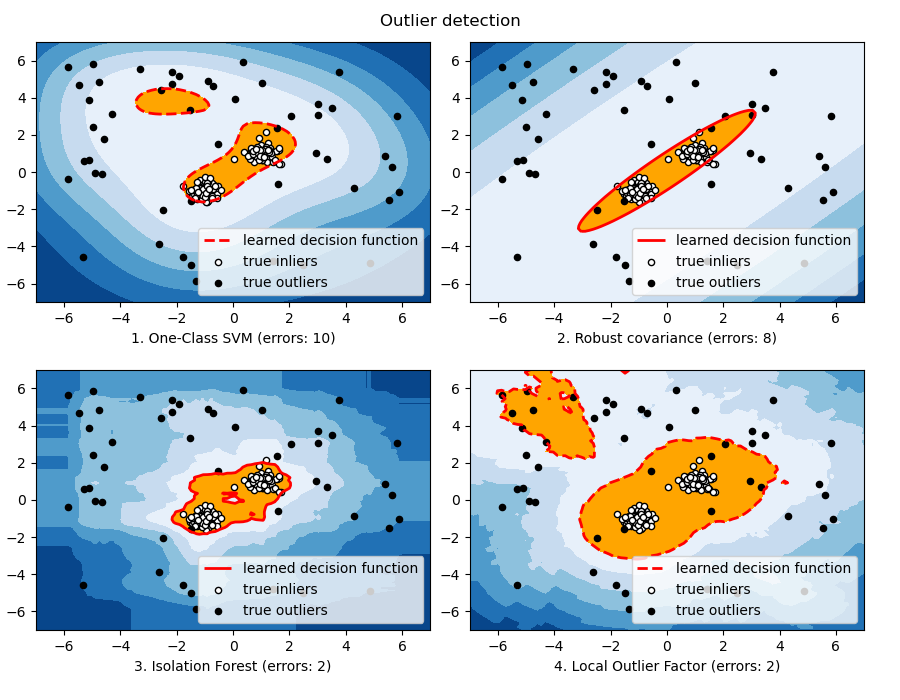
4.1.5 异常检测算法比较
import numpy as np
from scipy import stats
import matplotlib.pyplot as plt
import matplotlib.font_manager
from sklearn import svm
from sklearn.covariance import EllipticEnvelope
from sklearn.ensemble import IsolationForest
from sklearn.neighbors import LocalOutlierFactor
rng = np.random.RandomState(42)
# Example settings
n_samples = 200
outliers_fraction = 0.25
clusters_separation = [0, 1, 2]
# define two outlier detection tools to be compared
classifiers = {
"One-Class SVM": svm.OneClassSVM(nu=0.95 * outliers_fraction + 0.05,
kernel="rbf", gamma=0.1),
"Robust covariance": EllipticEnvelope(contamination=outliers_fraction),
"Isolation Forest": IsolationForest(max_samples=n_samples,
contamination=outliers_fraction,
random_state=rng),
"Local Outlier Factor": LocalOutlierFactor(
n_neighbors=35,
contamination=outliers_fraction)}
# Compare given classifiers under given settings
xx, yy = np.meshgrid(np.linspace(-7, 7, 100), np.linspace(-7, 7, 100))
n_inliers = int((1. - outliers_fraction) * n_samples)
n_outliers = int(outliers_fraction * n_samples)
ground_truth = np.ones(n_samples, dtype=int)
ground_truth[-n_outliers:] = -1
# Fit the problem with varying cluster separation
for i, offset in enumerate(clusters_separation):
np.random.seed(42)
# Data generation
X1 = 0.3 * np.random.randn(n_inliers // 2, 2) - offset
X2 = 0.3 * np.random.randn(n_inliers // 2, 2) + offset
X = np.r_[X1, X2]
# Add outliers
X = np.r_[X, np.random.uniform(low=-6, high=6, size=(n_outliers, 2))]
# Fit the model
plt.figure(figsize=(9, 7))
for i, (clf_name, clf) in enumerate(classifiers.items()):
# fit the data and tag outliers
if clf_name == "Local Outlier Factor":
y_pred = clf.fit_predict(X)
scores_pred = clf.negative_outlier_factor_
else:
clf.fit(X)
scores_pred = clf.decision_function(X)
y_pred = clf.predict(X)
# 选取预定的前25%的分数的分界线作为阈值
threshold = stats.scoreatpercentile(scores_pred, 100 * outliers_fraction)
# 计算误差
n_errors = (y_pred != ground_truth).sum()
# 绘制等高线
if clf_name == "Local Outlier Factor":
# decision_function is private for LOF
Z = clf._decision_function(np.c_[xx.ravel(), yy.ravel()])
else:
Z = clf.decision_function(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
subplot = plt.subplot(2, 2, i + 1)
subplot.contourf(xx, yy, Z, levels=np.linspace(Z.min(), threshold, 7),
cmap=plt.cm.Blues_r)
# 用红线画阈值边界
a = subplot.contour(xx, yy, Z, levels=[threshold],
linewidths=2, colors='red')
# 用橙色填充阈值区域内的背景
subplot.contourf(xx, yy, Z, levels=[threshold, Z.max()],
colors='orange')
b = subplot.scatter(X[:-n_outliers, 0], X[:-n_outliers, 1], c='white',
s=20, edgecolor='k')
c = subplot.scatter(X[-n_outliers:, 0], X[-n_outliers:, 1], c='black',
s=20, edgecolor='k')
subplot.axis('tight')
subplot.legend(
[a.collections[0], b, c],
['learned decision function', 'true inliers', 'true outliers'],
prop=matplotlib.font_manager.FontProperties(size=10),
loc='lower right')
subplot.set_xlabel("%d. %s (errors: %d)" % (i + 1, clf_name, n_errors))
subplot.set_xlim((-7, 7))
subplot.set_ylim((-7, 7))
plt.subplots_adjust(0.04, 0.1, 0.96, 0.94, 0.1, 0.26)
plt.suptitle("Outlier detection")
plt.show()
4.2 PyOD
from pyod.models.knn import KNN # imprt kNN分类器
from pyod.utils.data import generate_data
from pyod.utils.data import evaluate_print
from pyod.utils.example import visualize
contamination = 0.1 # percentage of outliers
n_train = 200 # number of training points
n_test = 100 # number of testing points
X_train, y_train, X_test, y_test = generate_data(
n_train=n_train, n_test=n_test, contamination=contamination)
# 训练一个kNN检测器
clf_name = 'kNN'
clf = KNN() # 初始化检测器clf
clf.fit(X_train) # 使用X_train训练检测器clf
# 返回训练数据X_train上的异常标签和异常分值
y_train_pred = clf.labels_ # 返回训练数据上的分类标签 (0: 正常值, 1: 异常值)
y_train_scores = clf.decision_scores_ # 返回训练数据上的异常值 (分值越大越异常)
# 用训练好的clf来预测未知数据中的异常值
y_test_pred = clf.predict(X_test) # 返回未知数据上的分类标签 (0: 正常值, 1: 异常值)
y_test_scores = clf.decision_function(X_test) # 返回未知数据上的异常值 (分值越大越异常)
# 评估预测结果
print("\nOn Test Data:")
evaluate_print(clf_name, y_test, y_test_scores)
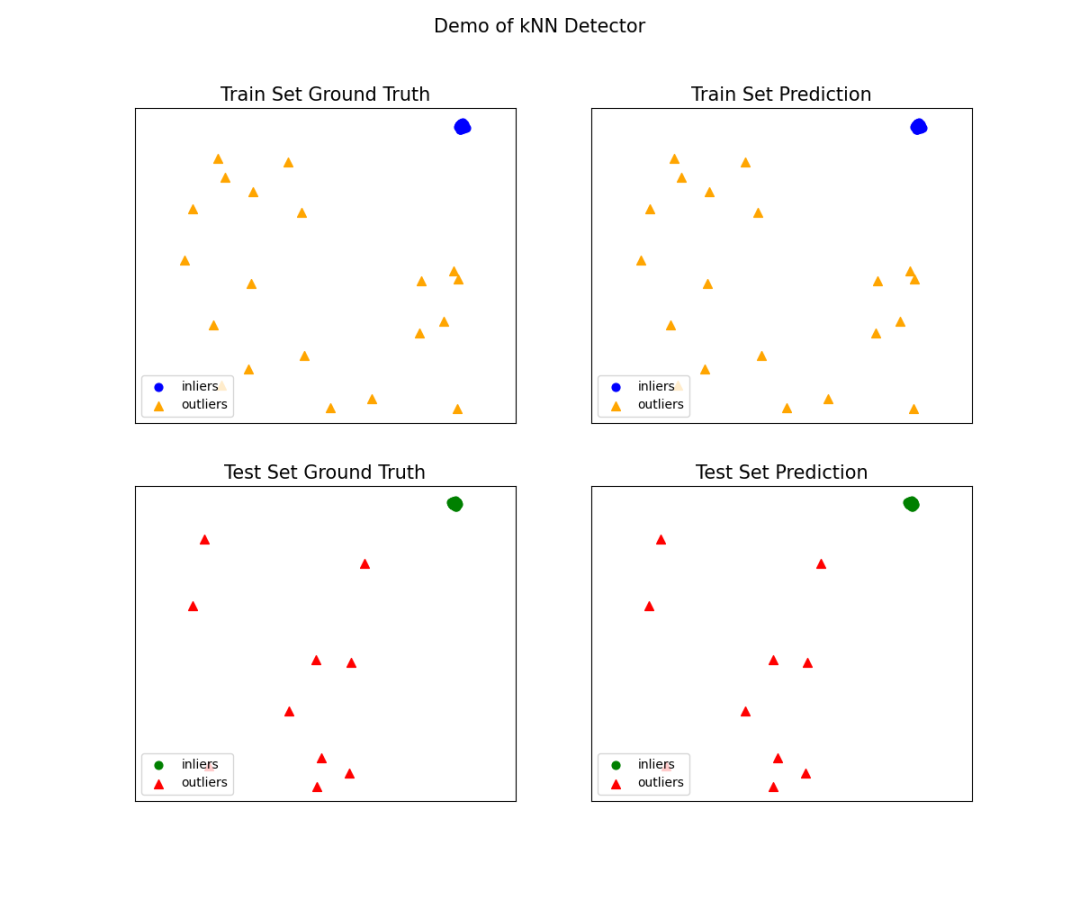
# 可视化
visualize(clf_name, X_train, y_train, X_test, y_test, y_train_pred,
y_test_pred, show_figure=True, save_figure=False)